.Decoding Neuronal Diversity: Study Illuminates Key RNA Edits in Synapse Proteins
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
https://www.facebook.com/junggoo.lee.54
.Decoding Neuronal Diversity: Study Illuminates Key RNA Edits in Synapse Proteins
신경 다양성 해독: 시냅스 단백질의 주요 RNA 편집을 밝히는 연구
주제:세포 생물학와 함께신경 과학RNA 작성자 DAVID ORENSTEIN, PICOWER 학습 및 기억 연구소 2023년 12월 16일 뇌 뉴런 매핑 아트 Drosophila 초파리의 뉴런은 뉴런 통신의 다양성을 이해하기 위해 Picower 학습 및 기억 연구소에서 연구되었습니다. 그들은 복합체인 단백질이 신경 전달 물질 방출을 조절하는 데 중요한 역할을 한다는 것을 발견했습니다. 이 연구는 콤플렉스의 RNA 편집으로 인해 다양한 버전의 단백질이 생성되어 뉴런이 통신하고 시냅스를 성장시키는 방식에 영향을 미치는 것으로 나타났습니다. 신용: SciTechDaily.com
뉴런은 확률적으로 최대 8가지 버전의 단백질 조절 신경전달 물질 방출을 생성하며, 이는 다른 세포와 통신하는 방식에 따라 달라질 수 있습니다. 뉴런은 말하는 사람입니다. 그들은 각각 "시냅스" 접합에서 신경 전달 물질 화학 물질을 방출하여 동료 뉴런, 근육 또는 다른 세포와 통신하여 궁극적으로 감정에서 동작에 이르는 기능을 생성합니다. 그러나 동일한 유형의 뉴런이라도 대화 스타일이 다를 수 있습니다.
Picower Institute for Learning and Memory의 신경생물학자들이 Cell Reports 저널에 게재한 새로운 공개 액세스 연구에서는 다음을 설명하는 데 도움이 될 수 있는 분자 메커니즘을 강조합니다. 신경 담론의 미묘한 다양성 과학자들은 초파리 초파리의 근육을 제어하는 뉴런에서 발견을 했습니다. 이 세포는 신경전달물질인 글루타메이트의 방출을 통한 의사소통을 포함하여 사람과 다른 동물의 뉴런에 공통된 많은 기본 특성을 나타내기 때문에 신경과학의 모델입니다.
뉴런이 이 중요한 과정을 조절하는 방법을 연구하는 MIT 생물학, 뇌 및 인지과학부의 Menicon 교수인 Troy Littleton 연구실에서 연구원들은 개별 뉴런의 방출 패턴이 다양하다는 것을 자주 볼 수 있습니다. 어떤 사람들은 다른 사람들보다 더 많이 "말"합니다.
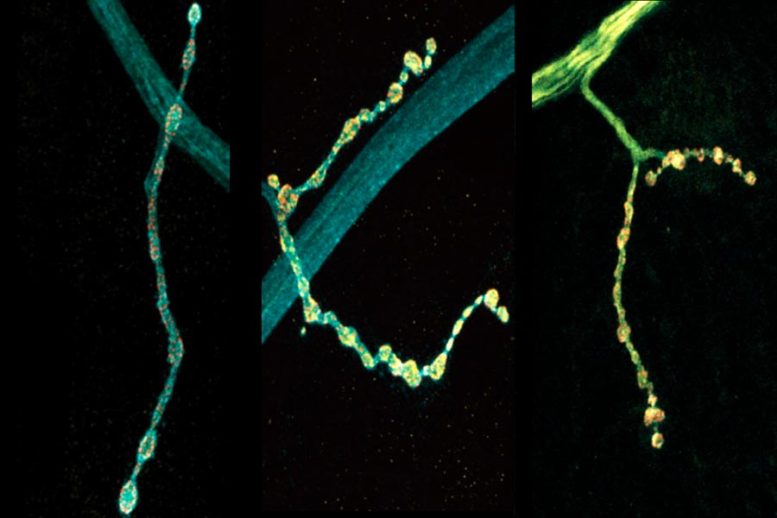
복합체 단백질 분포 운동 뉴런 신경 전달 물질의 방출을 통해 뉴런이 의사소통하는 방식을 조절하는 주요 단백질에 대한 새로운 연구에서 과학자들은 RNA 편집이 단백질의 분포와 성능에 어떻게 영향을 미치는지 추적했습니다. 여기에서 컴플렉스인(노란색)의 세 가지 다른 편집으로 인해 운동 뉴런의 세그먼트에서 단백질의 분포가 다르고 기능도 달라졌습니다. 왼쪽 패널은 편집되지 않은 컴플렉스의 분포를 보여주고 오른쪽 두 패널은 두 개의 서로 다른 편집된 변형의 분포를 보여줍니다. 크레딧: Littleton Lab/Picower 연구소
신경 통신에서 컴플렉스신의 역할 10년이 넘는 연구를 통해 Littleton의 연구실에서는 컴플렉스신이라는 단백질이 자발적인 글루타메이트 대화를 억제하는 역할을 한다는 사실을 보여주었습니다 . 이는 근육이 움직이는 것을 시뮬레이션하기 위해 뉴런이 기능적 이유로 신경전달물질을 필요로 할 때 신경전달물질의 공급을 보존하기 위해 시냅스막에서 글루타메이트가 채워진 소포의 융합을 단속합니다. 연구실의 연구에서는 초파리에서 두 가지 종류의 컴플렉스(포유류에는 4개가 있음)가 식별 되었으며 드물지만 강력한 7B 스플라이스 형태의 클램핑 효과가 다음과 같은 것으로 나타났습니다.
인산화라고 불리는 분자 과정에 의해 조절됩니다. 훨씬 더 풍부한 7A 버전이 어떻게 규제되는지는 알려지지 않았지만 과학자들은 RNA가 DNA에서 전사된다는 것을 보여주었습니다. 단백질 형성을 지시하는 것은 때때로 ADAR라는 효소에 의해 세포 내에서 편집됩니다. Elizabeth Brija PhD '23이 이끄는 Littleton 팀의 새로운 연구에서 연구소는 복합체 7A의 RNA 편집이 글루타메이트 방출을 조절하는 방법에 영향을 미치는지 여부를 조사했습니다. 그녀가 발견한 것은 놀라웠습니다.
-콤플렉스 7A의 RNA 편집은 단백질이 글루타메이트 방출을 얼마나 잘 방지하는지에 중요한 영향을 미칠 뿐만 아니라 최대 8개의 서로 다른 단백질 버전을 확률론적으로 혼합하고 일치시킬 수 있기 때문에 개별 뉴런마다 크게 다를 수 있습니다. 일부 편집은 평균적으로 다른 것보다 훨씬 더 흔했지만, 팀이 조사한 200개의 뉴런 중 96%에는 적어도 일부 편집이 있었으며, 이는 C-말단이라고 불리는 단백질 끝의 구조에 영향을 미쳤습니다. 이러한 구조적 변화의 결과 중 일부를 테스트하기 위한 실험에서는 7A 편집의 다양한 복합체가 다양한 시냅스에서 측정할 수 있는 전류 수준에 극적으로 영향을 미칠 수 있음을 보여주었습니다. 이러한 다양한 활동 수준은 뉴런이 근육과 만드는 시냅스의 성장에도 영향을 미칠 수 있습니다.
-따라서 단백질의 RNA 편집은 각 뉴런에 미세한 수준의 의사소통 제어 기능을 부여할 수 있습니다. Littleton은 "이것이 신경계에 제공하는 것은 동일한 전사체를 취하고 다양한 RNA 전사체를 대안적으로 편집함으로써 이들 뉴런이 다르게 행동할 수 있다는 것"이라고 말했습니다.
-범위 확장: 다른 단백질 편집 또한 Littleton과 Brija 팀은 시냅신 및 Syx1A와 같이 시냅스 글루타메이트 방출과 관련된 다른 주요 단백질도 때로는 동일한 뉴런 집단에서 상당히 다른 수준에서 편집된다는 사실을 발견했습니다.
-이는 시냅스 통신의 다른 측면도 조정할 수 있음을 시사합니다. Brija, Littleton 및 동료들은 "이러한 메커니즘은 신경 출력의 여러 기능을 변경하는 강력한 방법이 될 것입니다."라고 썼습니다. 팀은 200개의 운동 뉴런의 핵과 세포체에서 RNA를 꼼꼼하게 추출하고 서열을 분석하여 다양한 편집 수준을 추적했습니다.
이 연구는 C-말단에 있는 2개의 아미노산을 암호화하는 3개의 아데노신 뉴클레오티드 중 하나가 다른 것으로 교체되어 8개의 아데노신 뉴클레오티드가 생성될 수 있음을 보여주기에 충분할 정도로 풍부한 데이터 세트를 산출했습니다. 다양한 버전의 단백질. 평균 뉴런에서는 7A 복합체의 거의 대부분이 편집되지 않은 반면, 7개의 편집된 버전은 매우 다양한 빈도 수준으로 나머지를 구성했습니다.
일부 다른 버전의 기능적 결과를 조사하기 위해 팀은 컴플렉스인을 제거한 다음 편집되지 않은 버전 또는 두 가지 다른 편집 버전을 다시 추가하여 파리를 "구출"했습니다. 실험에서는 편집된 두 단백질 사이에 뚜렷한 대조가 나타났습니다. 더 일반적으로 발생하는 하나는 편집되지 않은 컴플렉스인보다 덜 효과적인 클램프로 입증되어 자발적인 글루타메이트 방출과 전류 상승을 거의 방지하지 못합니다.
다른 하나는 편집되지 않은 버전보다 클램핑에 더 효과적이며 글루타메이트 방출과 시냅스 출력을 단단히 유지하는 것으로 나타났습니다. 그리고 편집된 두 버전 모두 시냅스에서 벗어나 세포체에서 뻗어 있는 긴 가지인 뉴런의 축삭으로 표류하는 경향을 보인 반면, 잘 고정된 버전은 시냅스의 과도한 성장을 방지한 반면 제대로 고정하지 않은 버전은 시냅스의 과도한 성장을 방지했습니다.
빈약한 연석. 여러 에디션이 뉴런에 존재하는 경우가 많기 때문에 Brija와 팀은 편집되지 않은 컴플렉스인과 약한 클램핑 에디션을 조합하여 컴플렉스가 없는 초파리를 "구출"하는 실험 세트를 한 번 더 수행했습니다. 그 결과는 두 가지가 혼합된 것이었습니다. 즉, 약한 클램핑 버전만 사용한 경우보다 자발적인 글루타메이트 방출이 감소했습니다. 연구 결과는 각 버전이 잠재적으로 글루타메이트 방출을 미세 조정할 수 있을 뿐만 아니라 이들 간의 조합이 조합 방식으로 작용할 수 있음을 시사합니다.
참조: Elizabeth A. Brija, Zhuo Guan, Suresh K. Jetti 및 J. Troy Littleton의 "단일 뉴런 내 Complexin C-말단의 확률론적 RNA 편집은 신경 전달 물질 방출을 조절합니다.", 2023년 9월 17일, DOI: 10.1016/j.celrep.2023.113152.셀 보고서 Brija와 Littleton 외에도 이 논문의 다른 저자는 Zhuo Guan과 Suresh Jetti입니다. 국립 보건원, JPB 재단, Picower 학습 및 기억 연구소가 연구를 지원했습니다.
===========================
메모 2312171652 나의 사고실험 qpeoms 스토리텔링
인간의 원시 DNA는 어떻게 진화 되었고 자손을 어떻게 퍼뜨렸는지 생물학적 시나리오에는 qpeoms 시나리오 역사의 스토리텔링이 있다. 그것은 msbase.dna.rna.qpeoms.history가 있다. 허허.
여기서 msbase를 원시인 네안데르탈인의 신경계 msbase.dna를 가진다고 가정 한다면, 이 dna.rna 뉴런은 확률적으로 최대 8가지?버버버전. 아니지 아니여, vixxer 좌표계 버전이면 모를까!
실제로는 dna.rna 뉴런은 수천억 가지 버전의 단백질 조절 신경전달 물질 방출을 생성할 것이며, 이는 다른 세포와 통신하는 방식에 따라 쉽게 달라질 수도 있다. 그래서 우리가 육감적으로 위기일발을 모면하거나 기필코 목적을 이루거나 운명을 스스로 의지적으로 바꿀 수 있는거여. 허허.

-Neurons stochastically produce up to eight different versions of protein-regulated neurotransmitter release, which can vary depending on how they communicate with other cells. Neurons are talkers. They each release neurotransmitter chemicals at “synaptic” junctions to communicate with fellow neurons, muscles, or other cells, ultimately producing functions ranging from emotions to actions. However, even neurons of the same type can have different conversational styles.
-RNA editing of complex 7A not only has a significant impact on how well the protein prevents glutamate release, but can also vary greatly across individual neurons because up to eight different versions of the protein can be stochastically mixed and matched. Although some edits were, on average, much more common than others, 96% of the 200 neurons the team examined had at least some edits, which affected the structure of the end of the protein, called the C-terminus. Experiments to test some of the consequences of these structural changes showed that different complexes of 7A edits can dramatically affect the level of current that can be measured at different synapses. These different levels of activity can also affect the growth of the synapses that neurons make with muscles.
-Therefore, RNA editing of proteins can give each neuron fine-level control of communication. “What this gives the nervous system is that by taking the same transcript and making alternative edits to different RNA transcripts, these neurons can behave differently,” Littleton said.
-Expanding the scope: Editing other proteins Littleton and Brija's team also found that other key proteins involved in synaptic glutamate release, such as synapsins and Syx1A, were also edited, sometimes at significantly different levels in the same population of neurons.
===========================
Memo 2312171652 My thought experiment qpeoms storytelling
Biological scenarios include storytelling of qpeoms scenario history, such as how human primitive DNA evolved and how descendants were spread. It has msbase.dna.rna.qpeoms.history. haha.
Here, assuming that msbase has the nervous system msbase.dna of a primitive Neanderthal, this dna.rna neuron has a probability of up to 8 types. No, no, maybe it's the vixxer coordinate system version!
In reality, dna.rna neurons will produce hundreds of billions of versions of protein-regulated neurotransmitter release, which can easily vary depending on how they communicate with other cells. That's why we can avoid a crisis with intuition, achieve our goal without fail, or change our fate with our own will. haha.
Sample oms (standard)
b 0 a c f d 0000e0
0 0 0 a c 0 f00bde
0 c 0 f a b 000e0d
e 0 0 d 0 c 0b0fa0
f 0 0 0 e 0 b0dac0
d 0 f 0 0 0 cae0b0
0 b 0 0 0 f 0ead0c
0 d e b 0 0 ac000f
c e d 0 b a 00f000
a 0 b 0 0 e 0dc0f0
0 a c e 0 0 df000b
0 f 0 0 d 0 e0bc0a
sample qoms (standard)
0000000011=2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000
0010000001
sample pms (standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
0000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
Sample oss.base (standard)
zxdxybzyz
zxdzxezxz
xxbyyxzz
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

.A Neuroscience Milestone: Complete Cellular Mapping of a Mammalian Brain
신경과학 이정표: 포유류 뇌의 완전한 세포 매핑
주제:뇌국립 보건원신경 과학인기 있는 작성자 NIH/국립정신건강연구소 2023년 12월 14일 뇌 신호 매핑 아트
연구자들이 쥐의 뇌에 있는 3,200만 개 이상의 세포를 매핑하는 최초의 포유류 뇌의 포괄적인 세포 지도를 만들었습니다. NIH BRAIN Initiative의 일부인 이 지도책은 뇌 세포 유형과 연결에 대한 전례 없는 통찰력을 제공하여 인간 뇌에 대한 이해를 높이고 뇌 장애에 대한 새로운 치료법 개발을 돕습니다. 신용: SciTechDaily.com
3,200만 개 이상의 세포를 자세히 설명하는 전체 쥐 뇌를 매핑하는 획기적인 세포 지도는 인간 뇌에 대한 더 깊은 이해와 뇌 장애에 대한 정밀 치료법 개발의 길을 열어줍니다. 국제 연구팀이 최초로 포유류 뇌 전체의 완전한 세포 지도를 만들었습니다.
이 지도책은 쥐 뇌의 지도 역할을 하며 3,200만 개가 넘는 세포의 유형, 위치, 분자 정보를 설명하고 이들 세포 간의 연결에 대한 정보를 제공합니다. 쥐는 신경과학 연구에서 가장 일반적으로 사용되는 척추동물 실험 모델이며, 이 세포 지도는 세계에서 가장 강력한 컴퓨터라고 할 수 있는 인간의 뇌를 더 잘 이해할 수 있는 길을 열어줍니다. 셀 아틀라스는 또한 뇌의 정신 및 신경 장애가 있는 사람들을 위한 차세대 정밀 치료법 개발의 토대를 마련합니다.
이 연구 결과는 국립 보건원 혁신적인 신경 기술 발전을 통한 뇌 연구® 이니셔티브의 자금 지원을 받았습니다. 또는 The BRAIN Initiative®에 게재되었으며 Nature에 게재된 10개 논문 모음에 게재되었습니다. "마우스 아틀라스는 포유동물 뇌 세포의 복잡한 네트워크에 전례 없는 초점을 맞춰 연구자들에게 인간의 뇌 기능과 질병을 이해하는 데 필요한 세부 정보를 제공했습니다."라고 이사인 Joshua A. Gordon, M.D., Ph.D.는 말했습니다.
국립보건원 산하 국립정신건강연구소 마우스 뇌의 상세한 매핑 세포 지도책은 마우스 뇌의 각 영역에 있는 세포 유형과 해당 영역 내 조직을 설명합니다. 이러한 구조적 정보 외에도 세포 지도책은 세포의 전사체에 대한 놀라울 정도로 상세한 카탈로그를 제공합니다. 즉, 단백질 및 기타 세포 산물을 만드는 지침이 포함된 세포 내 유전자 판독의 전체 세트입니다. 아틀라스에 포함된 전사체 정보는 계층적으로 구성되어 뇌 내의 세포 클래스, 하위 클래스 및 수천 개의 개별 세포 클러스터를 자세히 설명합니다. 지도책은 또한 세포 후성유전체(세포의 DNA에 대한 화학적 변형 및 세포의 유전 정보가 표현되는 방식을 변경하는 염색체)의 특성을 나타냅니다.
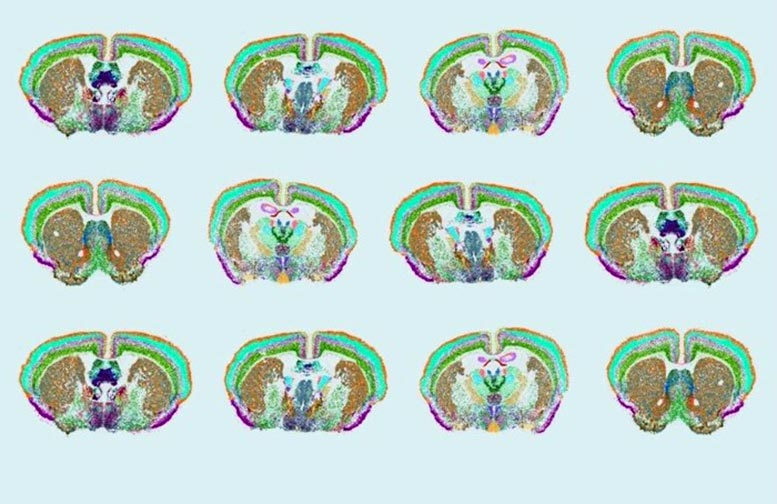
수천 개의 후성유전체 세포 유형과 다양한 뇌 세포 유형에 대한 수백만 개의 후보 유전자 조절 요소를 자세히 설명합니다. 마우스 뇌의 다양한 세포 유형의 공간 분포 마우스 뇌의 다양한 세포 유형의 공간적 분포. 여기서 MERFISH는 마우스 뇌에서 500개의 유전자를 측정하여 뇌 전체에 걸쳐 세포 유형의 복잡한 분포를 밝히는 데 사용되었습니다. 출처: Yao/van Velthoven/Zeng, Allen Institute
이 지도책에 포함된 구조적, 전사적, 후성유전학적 정보는 함께 마우스 뇌 전반에 걸쳐 세포 조직과 다양성에 대한 전례 없는 지도를 제공합니다. 지도책은 또한 다양한 세포에서 사용되는 신경전달물질과 신경펩티드, 그리고 뇌 내 세포 유형 간의 관계에 대한 설명을 제공합니다. 이 정보는 뇌의 여러 부분에서 화학적 신호가 어떻게 시작되고 전송되는지에 대한 자세한 청사진으로 사용될 수 있습니다.
이러한 전기 신호는 뇌 회로가 작동하는 방식과 뇌가 전반적으로 기능하는 방식에 대한 기초입니다. 선구적인 협업 노력과 미래 방향 NIH BRAIN Initiative의 이사인 John Ngai 박사는 “이 제품은 전례 없는 교차 협업의 힘을 보여주는 증거이며 보다 정밀한 뇌 치료를 위한 길을 열어줍니다.”라고 말했습니다. 이 컬렉션에 포함된 10개 연구 중 7개는 NIH BRAIN Initiative Cell Census Network(BICCN)을 통해 자금을 지원받고, 2개는 더 큰 규모의 NIH BRAIN Initiative를 통해 자금을 지원받습니다.
뇌의 세포 구성을 이해하기 위한 획기적인 교차 협력 노력인 BICCN의 핵심 목표는 뇌 세포의 위치, 발달 방식, 함께 작동하는 방식 및 세포의 포괄적인 목록을 개발하는 것입니다. 뇌 장애가 어떻게 발생하고 진행되며 가장 잘 치료되는지 더 잘 이해하기 위해 활동을 조절합니다. Ngai 박사는 “BICCN은 다학문적, 국제적 협력의 고유한 특성을 활용하여 이전에 다른 어떤 과학자 팀도 할 수 없었던 일을 성취할 수 있었습니다.”라고 말했습니다.
"이제 우리는 인간 뇌와 인간이 아닌 영장류 뇌의 세포 지도를 완성하는 다음 큰 단계를 밟을 준비가 되었습니다." BRAIN Initiative Cell Atlas Network(BICAN)은 포유류 뇌의 세포와 세포 기능을 이해하려는 NIH BRAIN Initiative 노력의 다음 단계입니다. BICAN은 다른 두 개의 대규모 프로젝트인 BRAIN Initiative Connectivity Across Scales 및 Armamentarium for Precision Brain Cell Access와 함께 행동의 회로 기반을 관리하는 기본 원칙을 밝히고 새로운 접근 방식을 알려 신경 과학 연구에 혁명을 일으키는 것을 목표로 하는 혁신적인 프로젝트입니다. 인간의 뇌 질환을 치료하는 데 사용됩니다.
참조: Zizhen Yao, Cindy T. J. van Velthoven, Michael Kunst, Meng Zhang, Delissa McMillen, 이창규, 정원, Jeff의 "전체 마우스 뇌에 있는 세포 유형의 고해상도 전사체 및 공간 지도책" Goldy, Aliya Abdelhak, Matthew Aitken, Katherine Baker, Pamela Baker, Eliza Barkan, Darren Bertagnolli, Ashwin Bhandiwad, Cameron Bielstein, Prajal Bishwakarma, Jazmin Campos, Daniel Carey, Tamara Casper, Anish Bhaswanth Chakka, Rushil Chakrabarty, Sakshi Chavan, Min Chen , 마이클 클락, 제니 클로즈, 커스틴 크라이튼, 스콧 다니엘, 피터 디발렌틴, 팀 돌베어, 로렌 엘링우드, 엘리샤 피아베인, 티모시 플리스, 제임스 지, 제임스 거스텐버거, 알렉산드라 글랜던, 제시카 글로, 조슈아 굴드, 제임스 그레이, 네이선 길포드, 주니타 구즈만, 다니엘 허쉬스타인, 윈디 호, 마커스 후퍼, 마이크 황, 매디 허프, 켈리 진, 매튜 크롤, 케이넌 라티아, 아리엘 레온, 수 리, 브라이언 롱, 잭 매디건, 제시카 말로이, 조슬린 말론, 조이 말처, 나오미 마틴, 레이첼 맥큐, 라이언 맥긴티, 니콜라스 메이, 호세 멜코르, 엠마 메이어디어크스, 타일러 몰렌코프, 스카일러 문스맨, 툭 응이 응우옌, 스벤 오토, 트랑탄 팜, 크리스틴 리모린, 오거스틴 루이스, 레이먼드 산체스, 레인 소여, 나디야 샤포발로바, 노아 셰퍼드, 클리프 Slaughterbeck, Josef Sulc, Michael Tieu, Amy Torkelson, Herman Tung, Nasmil Valera Cuevas, Shane Vance, Katherine Wadhwani, Katelyn Ward, Boaz Levi, Colin Farrell, Rob Young, Brian Staats, Ming-Qiang Michael Wang, Carol L. Thompson, Shoaib Mufti, Chelsea M. Pagan, Lauren Kruse, Nick Dee, Susan M. Sunkin, Luke Esposito, Michael J. Hawrylycz, Jack Waters, Lydia Ng, Kimberly Smith, Bosiljka Tasic, Xiaowei Zhuang 및 Hongkui Zeng, 2023년 12월 13일, < /span>DOI: 10.1038/s41586-023-06812-z.자연
https://scitechdaily.com/a-neuroscience-milestone-complete-cellular-mapping-of-a-mammalian-brain/
댓글