.New single-cell RNA-sequencing method has potential to become universal tool of choice
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
http://jk0620.tripod.com
https://www.facebook.com/junggoo.lee.9

.New single-cell RNA-sequencing method has potential to become universal tool of choice
새로운 단일 세포 RNA 시퀀싱 방법은 보편적인 선택 도구가 될 가능성이 있습니다
분자 임상 안과 연구소 바젤 pre-mRNA의 헤어핀 루프. 강조 표시된 것은 핵염기(녹색)와 리보스-인산 백본(파란색)입니다. 이것은 스스로 접히는 RNA의 단일 가닥이라는 점에 유의하십시오. 크레딧: Vossman/ Wikipedia MAY 30, 2022
-IOB(Institute of Molecular and Clinical Ophthalmology Basel)에서 개발한 새로운 단일 세포 RNA 시퀀싱 프로토콜을 사용하면 기존 방법보다 세포당 훨씬 많은 수의 유전자를 검출할 수 있습니다. 또한 더 빠르고 저렴하며 더 민감합니다. 새로운 방법은 현재 Nature Biotechnology 에 게재되었습니다 . 단일 세포 RNA 시퀀싱(scRNA-seq)은 세포에서 어떤 유전자가 활성화되고 전사 수준이 어느 정도인지 보여줍니다. 이를 통해 개별 세포의 생물학을 심층적으로 평가하고 질병 을 나타낼 수 있는 변화를 감지할 수 있습니다 .
scRNA-seq는 발달 생물학, 신경학, 종양학, 면역학, 심혈관 연구 및 전염병을 포함한 여러 학문 분야에서 널리 사용되고 있습니다. 인구 이질성을 연구하고 관심 있는 소수 하위 집단을 식별하며 개별 세포의 고유한 특성을 발견하는 데 중요합니다. 응용 프로그램은 안과를 훨씬 능가합니다. 단일 세포 시퀀싱은 정상 및 병든 세포 의 새로운 생식계열 돌연변이 및 체세포 돌연변이 분석에 널리 사용됩니다.
예를 들어 암세포에서. IOB에서 Novartis Institutes for BioMedical Research의 과학자들과 함께 개발한 프로토콜은 고해상도의 희귀 세포 집단 분석이 필요한 모든 질병 모델을 연구하는 데 사용할 수 있습니다. IOB 단일 세포 유전체학 플랫폼의 책임자이자 이 논문의 수석 저자인 Simone Picelli는 "우리의 모듈식 FLASH-seq 프로토콜은 전례 없는 해상도로 세포 전사체의 스냅샷을 제공합니다. 이 방법은 소형화, 자동화 및 적응할 수 있습니다. 건강과 질병에 존재하는 유전자 isoform을 정의하는 데 도움이 됩니다. 또한 특히 질병, 발달 결함 또는 외부 요인으로 인한 교란 후 유전자 발현에 대한 훨씬 더 깊은 그림을 제공합니다. 게다가 설정도 쉽습니다. 실험실에서, 새로운 방법은 단 반나절 만에 시퀀싱 준비 라이브러리를 생성할 수 있습니다. 따라서 IOB의 연구원들은 FLASH-seq가 효율적이고 강력하며 모듈식이며 저렴하고 자동화 친화적인 전체 길이 scRNA-seq 프로토콜 을 찾을 때 선택 도구가 될 가능성이 있다고 믿습니다 .
추가 탐색 DisCo: 단일 세포 RNA 시퀀싱의 효율성 향상 추가 정보: Vincent Hahaut et al, FLASH-seq를 사용하여 빠르고 민감한 전체 길이 단일 세포 RNA 시퀀싱, Nature Biotechnology (2022). DOI: 10.1038/s41587-022-01312-3 저널 정보: 네이처 바이오테크놀로지
https://phys.org/news/2022-05-single-cell-rna-sequencing-method-potential-universal.html
=================
메모 2205310446 나의 사고살험 oms 스토리텔링
DNA는 샘플c.oss의 초기값 베이스 마방진과도 같다. 물론 인간의 30억개의 염기를 가진 대략 54,772차 마방진이 될 것이다. 이는 가로 세로 주대각선의 합이 동일한 유전자의 5만4천여 종류를 나타내는듯 하다.
이러한 DNA 베이스를 증식 시키려면 RNA.oss 원시모드가 절대적으로 필요하다. DNA원본을 가져다 복제판을 만들려면 원본의 반쪽을 가져다 나머지 재료를 찾아서 붙이는 경우도 있다. 이런 역할이 바로 생물학적 RNA인데 샘플c.oss는 원본자체를 더 크게 만든 것이고 동일한 복제물은 아니다. 하지만 원본자체의 종류수를 늘리여 DNA 유전자의 종류가 엄청나게 많다는 것을 알 수 있는 방법이 제공되기도 한다.
단순 복제가 아닌 확장 원본에서 RNA복제를 찾는다면 우리가 이전에서 본 원본 DNA에서의 유전자보다 더 세밀한 부분이 보일 수 있다.
고로, 샘플c.oss.rna을 통해 유전자의 디테일한 세부목록을 찾아낼 수도 있을 것이다. 허허.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):

-The new single-cell RNA sequencing protocol developed by the Institute of Molecular and Clinical Ophthalmology Basel (IOB) allows the detection of significantly higher numbers of genes per cell than conventional methods. It is also faster, cheaper and more sensitive. The new method is now published in Nature Biotechnology. Single-cell RNA sequencing (scRNA-seq) reveals which genes are active in the cell and at what level of transcription. This allows for in-depth assessment of the biology of individual cells and detects changes that may indicate disease.
Intermediary role from DNA to protein production
As is commonly known, DNA contains unique, unique genetic information for each individual. Genetic information can be said to be information about what kind of cells to make and how to construct it from internal organs to the overall appearance. After all, it is information to make the protein our body needs in the right place. For this, someone has to 'interpret' the information commanded by DNA and then play a role of 'transmitting' it to the place where the protein is made.
Material 1.
mRNA that copies genetic information from DNA and is brought out of the cell nucleus
It is RNA that serves as such a bridge. As an analogy, let's say you have a designer (DNA) who creates a new product. Then someone has to bring the blueprints to the factory. Engineers in the factory will order the necessary materials (amino acids) by looking at the drawings delivered, and when the materials arrive at the factory, they will assemble them according to the design drawings to create a finished product (protein).
If we go step by step, there is the RNA that delivers the blueprints of DNA to the factory. This RNA is called mRNA, after the first letter of messenger. There are also RNAs that are obtained from factory orders and delivered to technicians. It is called tRNA, meaning transfer. RNA that uses materials brought from tRNA to make a finished product according to design drawings is called rRNA. The 'r' in front is because the name of the factory, that is, the name of the organelle that makes proteins, is ribosome.
Why is RNA more unstable than DNA?
In addition, various types of RNA and their roles have been recently revealed one after another in life science research, but it seems that you only need to know this far. So let's see what is the difference between RNA and DNA. RNA stands for RiboNucleic Acid. Ribonucleic acid is a sugar molecule called 'ribose' with a phosphoric acid and a base attached to it. On the other hand, DNA contains deoxyribose, not ribose, although all other parts are identical.
Chemical structures of RNA (left) and DNA - provided by Dong-A Science Co., Ltd.
Chemical structures of RNA (left) and DNA - provided by Dong-A Science Co., Ltd.
If you look at the chemical structure, RNA or DNA are so similar that you can't tell them apart. At the position where the hydroxyl group (-OH) is attached to the ribose of RNA, the deoxyribose of DNA has a hydrogen (-H) that is deoxygenated. The name deoxyribose in DNA comes from ‘deoxy’, which means that oxygen is missing, before ribose.
This is where RNA and DNA make an important difference. The hydroxyl group (-OH) of RNA has the characteristic of being active in chemical reactions. Being able to respond well to others means being unstable in the sense that it produces a variety of results. Conversely, the fact that it does not react well with other elements like deoxyribose in DNA is stable in that it does not change.
====================
Memo 2205310446 My accidental murder oms storytelling
DNA is the same as the initial base magic square of sample c.oss. Of course, it would be roughly 54,772 order of magnitude magic square with 3 billion bases in humans. It seems that the sum of the horizontal, vertical and main diagonals represents about 54,000 types of the same gene.
RNA.oss native mode is absolutely necessary to propagate these DNA bases. If you want to make a clone by taking the original DNA, you may need to take half of the original, find the rest of the material, and attach it. This role is biological RNA, and the sample c.oss is a larger version of the original, not an identical copy. However, there is also a way to know that there are a huge number of types of DNA genes by increasing the number of original types.
If we look for RNA replication from an extended source rather than a simple copy, we can see more detailed parts than the gene in the original DNA we have seen before.
Therefore, it may be possible to find a detailed catalog of genes through the sample c.oss.rna. haha.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):

.Astronomers detect a new radio source of unknown origin
천문학자들은 출처를 알 수 없는 새로운 전파원을 감지합니다
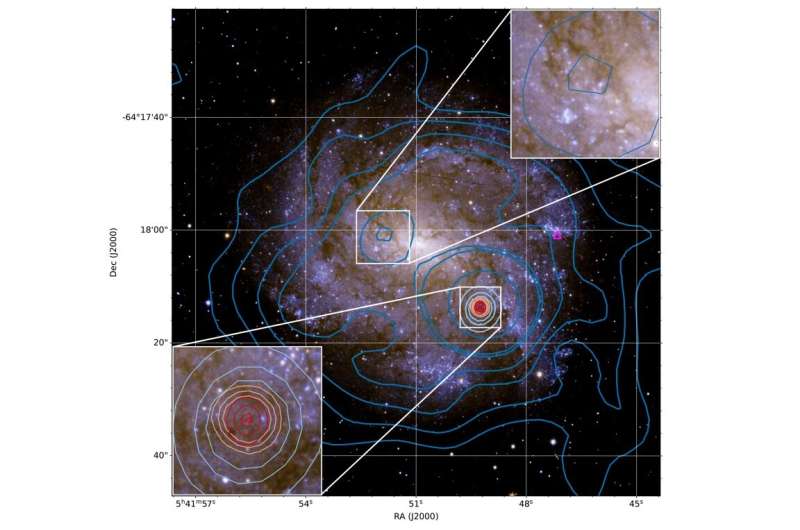
작성자: Tomasz Nowakowski, Phys.org ASKAP 및 ATCA 등고선이 오버레이된 NGC 2082의 3색 HST 이미지. 왼쪽 하단의 삽입 이미지는 J054149.24–641813.7의 확대를 제공하여 광학 대응물이 없음을 보여줍니다. 출처: Balzan et al., 2022.MAY 31, 2022 REPORT
-NGC 2082로 알려진 나선 은하의 전파 연속체 관측 중 호주 천문학자들은 J054149.24–641813.7이라는 명칭을 받은 신비하고 밝고 조밀한 전파원을 발견했습니다. 이 출처의 기원과 성격은 알려지지 않았으며 추가 조사가 필요합니다. 이 발견은 5월 23일 arXiv 인쇄 전 리포지토리에 게시된 논문에 보고되었습니다. 일반적으로 전파원은 상대적으로 많은 양의 전파를 방출하는 우주의 다양한 물체입니다. 그러한 방출의 가장 강력한 소스 중에는 펄서, 특정 성운, 퀘이사 및 전파 은하 가 있습니다. 이제 호주 웨스턴 시드니 대학의 조엘 발잔(Joel Balzan)이 이끄는 천문학자 팀이 새로운 전파원의 발견을 보고했는데, 그 정체는 여전히 불확실합니다.
ASKAP(Australian Square Kilometer Array Pathfinder), ATCA(Australian Telescope Compact Array) 및 Parkes 전파 망원경을 사용하여 NGC 2082를 관찰하는 동안 은하 중심에서 20 arcseconds 위치에 있는 강력한 전파 소스를 식별했습니다. NGC 2082는 지구에서 약 6000만 광년 떨어져 있는 황새자리에 있는 G형 나선은하로 지름은 약 3만3000광년이다. "우리는 888MHz에서 9,000MHz까지 ASKAP, ATCA 및 Parkes 망원경을 사용하여 NGC 2082의 전파 연속체 관측을 제시합니다. 이 가까운 나선 은하 의 중심에서 약 20 arcsec 떨어진 곳 에서 우리는 밝고 작은 전파원 J054149.24–641813.7을 발견했습니다. 출처를 알 수 없다"고 연구진은 논문에 썼다.
연구에 따르면 888MHz에서 J054149.24–641813.7의 전파 광도는 129EW/Hz 수준이며 전파 스펙트럼 지수(약 0.02)가 평평한 것으로 나타났습니다. 천문학자들에 따르면 이것은 J054149.24–641813.7이 초신성 잔해(SNR) 또는 펄서일 수 있다는 시나리오에 반대하며, 이는 소스가 열 기원일 수 있음을 시사합니다. 연구원들은 J054149.24–641813.7의 컴팩트한 특성과 NGC 2082 외곽의 위치가 일부 FRB( 고속 무선 버스트 )를 연상시킨다는 점에 주목했습니다. 그러나 결과는 J054149.24–641813.7이 FRB 전구가 내장된 영구 무선 소스가 되기에 충분히 밝지 않을 수 있음을 시사합니다. 천문학자들은 J054149.24–641813.7이 준 항성 물체(QSO, 퀘이사), 전파은하 또는 활성은하핵(AGN)과 같은 은하외 배경 소스일 가능성이 가장 높다고 결론지었습니다. 그들은 5,500 및 9,000MHz에서 다소 약한 편파와 함께 평평한 스펙트럼 지수가 이 가설을 뒷받침한다고 덧붙였습니다. 그러나 현재 NGC 2082에 대한 고해상도 중성 원자 수소(HI) 흡수 데이터가 없으므로 이 가정을 확인할 수 있습니다. "우리는 NGC 2082 배후에서 그러한 근원을 찾을 확률이 P = 1.2%라는 것을 발견했고, J054149.24–641813.7의 가장 가능성 있는 기원은 배경 퀘이사 또는 전파 은하라고 결론지었습니다."
추가 탐색 천문학자들은 새로운 은하 외 원형 전파원을 발견합니다. 추가 정보: Joel CF Balzan et al, A Radio Continuum Study of NGC 2082. arXiv:2205.11144v1 [astro-ph.GA], arxiv.org/abs/2205.11144
https://phys.org/news/2022-05-astronomers-radio-source-unknown.html
===================
메모 2206010407 나의 사고실험 oms 스토리텔링
심우주에 관한 샘플a.oms는 smola.mser의 열림과 닫힘의 문제인듯하다. 이에 따라 소스가 불분명한 전파원이 나타날 수도 있는 점이다. 허허. 닫힌 심우주에서 안보이던 전파원이 열린 심우주에서 보일 것이고 다시 닫히면 사라지는 이런 식의 시스템은 오직 oms.블랙홀 vixer에 의해 좌우된다.
심우주 smola.mser의 개폐성은 블랙홀 vixer와 직접적으로 관련돼 있어 보인다. 허허.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):

-During observations of the radio continuum of the spiral galaxy known as -NGC 2082, Australian astronomers discovered a mysterious, bright and dense radio source, designated J054149.24–641813.7. The origin and nature of this source are unknown and require further investigation. This finding was reported in a paper published May 23rd in the arXiv pre-print repository. In general, radio sources are various objects in space that emit relatively large amounts of radio waves. Among the most powerful sources of such emissions are pulsars, certain nebulae, quasars, and radio galaxies. Now, a team of astronomers led by Joel Balzan of the University of Western Sydney in Australia has reported the discovery of a new radio source, the identity of which remains uncertain.
=====================
memo 2206010407 my thought experiment oms storytelling
Sample a.oms about the deep universe seems to be a matter of opening and closing smola.mser. Accordingly, a radio wave source with an unknown source may appear. haha. A radio source that was invisible in a closed deep universe would be visible in an open deep universe and disappear when closed again. This type of system is governed only by the oms.black hole vixer.
The openability of the deep universe smola.mser seems to be directly related to the black hole vixer. haha.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):
댓글