.Scientists Have Spotted the Farthest Galaxy Ever – It May Be Home to the Oldest Stars in the Universe
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
http://jk0620.tripod.com
https://www.facebook.com/junggoo.lee.9
.Scientists Have Spotted the Farthest Galaxy Ever – It May Be Home to the Oldest Stars in the Universe
과학자들은 지금까지 가장 먼 은하를 발견했습니다 – 그것은 우주에서 가장 오래된 별의 고향일 수 있습니다

주제:천문학천체물리학블랙홀하버드-스미소니언 천체 물리학 센터 2022년 4월 7일 Harvard -SMITHSONIAN 천체 물리학 센터 작성 갤럭시 HD1 빨간색 물체인 HD1은 확대 이미지의 중앙에 나타납니다. 크레딧: Harikane et al.
빅뱅 이후 약 3억 년 만에 빛나는 이 별은 우주에서 가장 오래된 별이나 초대질량 블랙홀의 고향일 수 있습니다 . 천체 물리학 센터의 연구원을 포함한 국제 천문학 팀 | Harvard & Smithsonian은 역사상 가장 먼 천체인 은하를 발견했습니다. HD1이라는 이름의 은하 후보는 약 135억 광년 떨어져 있으며 오늘(2022년 4월 7일) 천체 물리학 저널 에 설명되어 있습니다 .
The Monthly Notices of the Royal Astronomical Society Letters에 게재된 첨부 논문에서 과학자들은 은하가 정확히 무엇인지 추측하기 시작했습니다. 팀은 두 가지 아이디어를 제안합니다. HD1은 놀라운 속도로 별을 형성하고 있을 수 있으며 지금까지 한 번도 관찰된 적이 없는 우주 최초의 별인 인구 III 별의 고향일 수도 있습니다. 또는 HD1은 우리 태양 질량의 약 1억 배에 달하는 초대질량 블랙홀을 포함할 수 있습니다.
MNRAS 연구의 주저자이자 ApJ에 대한 발견 논문의 공동 저자이자 천체 물리학 센터의 천문학자인 Fabio Pacucci는 "지금까지 멀리 있는 소스의 특성에 대한 질문에 답하는 것은 어려울 수 있습니다."라고 말합니다 . “그 배는 강풍과 짙은 안개 한가운데에 머나먼 해안에 있으면서 펄럭이는 깃발로 배의 국적을 짐작하는 것과 같습니다. 국기의 일부 색상과 모양을 볼 수 있지만 전체를 볼 수는 없습니다. 그것은 궁극적으로 분석과 타당하지 않은 시나리오의 배제의 긴 게임입니다. HD1은 자외선에서 매우 밝습니다. 이것을 설명하기 위해 "일부 에너지 과정이 그곳에서 일어나고 있거나 더 좋게는 수십억 년 전에 일어났습니다"라고 Pacucci는 말합니다.
처음에 연구원들은 HD1이 빠른 속도로 별을 생성하는 표준 항성 폭발 은하라고 가정했습니다. 그러나 HD1이 생성하는 별의 수를 계산한 후 그들은 "놀라운 비율 - HD1은 매년 100개 이상의 별을 생성할 것입니다. 이것은 우리가 이 은하에 대해 기대하는 것보다 최소 10배 더 높습니다.” 그 때 팀은 HD1이 평범한 일상의 별을 형성하지 않을 수 있다고 의심하기 시작했습니다.
Pacucci는 "우주에서 형성된 최초의 별들은 현대의 별보다 더 무겁고 더 밝으며 더 뜨겁습니다."라고 말합니다. “HD1에서 생성된 별이 이 첫 번째 별, 즉 인구 III의 별이라고 가정하면 그 속성을 더 쉽게 설명할 수 있습니다. 사실, Population III 별은 일반 별보다 더 많은 자외선을 생성할 수 있어 HD1의 극자외선 광도를 명확히 할 수 있습니다.” 그러나 초대질량 블랙홀은 HD1의 극도의 광도를 설명할 수도 있습니다. 엄청난 양의 가스를 삼키면서 블랙홀 주변 지역에서 고에너지 광자가 방출될 수 있습니다. 만약 그렇다면 현재의 기록 보유자에 비해 빅뱅에 훨씬 더 가까운 시간에 관측된 인류에게 알려진 가장 오래된 초거대질량 블랙홀이 될 것입니다.
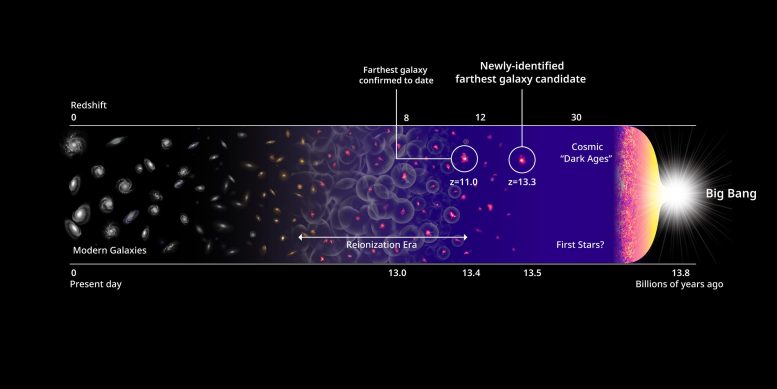
타임라인 오브 유니버스의 갤럭시 HD1 타임라인은 최초의 은하 후보와 우주의 역사를 표시합니다. 출처: Harikane et al., NASA, EST 및 P. Oesch/Yale
"HD1은 초기 우주의 분만실에 있는 거대한 아기를 나타냅니다."라고 MNRAS 연구의 공동 저자이자 천체 물리학 센터의 천문학자인 Avi Loeb가 말했습니다. "그것은 기록상 가장 높은 퀘이사 적색편이를 거의 2배 정도 깨는 놀라운 위업입니다." HD1은 스바루 망원경, 비스타 망원경, 영국 적외선 망원경, 스피처 우주 망원경으로 1,200시간 이상의 관찰 시간 끝에 발견되었습니다.
"700,000개 이상의 물체 중에서 HD1을 찾는 것은 매우 힘든 작업이었습니다."라고 도쿄 대학의 천문학자이자 은하계를 발견한 Yuichi Harikane은 말합니다. "HD1의 붉은 색은 135억 광년 떨어진 은하의 예상되는 특성과 놀라울 정도로 잘 맞아 발견했을 때 소름이 돋았습니다." 그런 다음 팀은 ALMA (Atacama Large Millimeter/submillimeter Array)를 사용하여 추적 관찰을 수행 하여 현재 가장 먼 은하에 대한 기록 보유자인 GN-z11보다 1억 광년 더 먼 거리를 확인했습니다. 연구팀은 James Webb 우주 망원경 을 사용하여 곧 다시 HD1을 관찰하여 지구로부터의 거리를 확인할 것입니다. 현재 계산이 맞다면 HD1은 지금까지 기록된 은하 중 가장 멀리 있고 가장 오래된 은하가 될 것입니다.
동일한 관찰을 통해 팀은 HD1의 정체성을 더 깊이 파고들고 그들의 이론 중 하나가 올바른지 확인할 수 있습니다. "빅뱅 이후 수억 년 후에 형성된 HD1의 블랙홀은 전례 없는 속도로 거대한 씨앗에서 자라났음에 틀림없습니다."라고 Loeb은 말합니다. "다시 한 번, 자연은 우리보다 더 상상력이 풍부한 것 같습니다."
참조: “A Search for H-Dropout Lyman Break Galaxies at z~12-16” by Yuichi Harikane, Akio K. Inoue, Ken Mawatari, Takuya Hashimoto, Satoshi Yamanaka, Yoshinobu Fudamoto, Hiroshi Matsuo, Yoichi Tamura, Pratika Dayal, LY Aaron , Anne Hutter, Fabio Pacucci, Yuma Sugahara and Anton M. Koekemoer, 7 April 2022, The Astrophysical Journal . DOI: arXiv:2112.09141 "새롭게 발견된 z∼13 드롭아웃 소스는 항성 폭발 은하인가 퀘이사인가?" Fabio Pacucci, Pratika Dayal, Yuichi Harikane, Akio K. Inoue 및 Abraham Loeb, 2022년 4월 7일, . DOI: arXiv:2201.00823 천체 물리학 센터 소개 | 하버드 & 스미소니언 천체 물리학 센터 | Harvard & Smithsonian은 Harvard와 Smithsonian의 공동 작업으로 우주의 본질에 대한 인류의 가장 큰 미해결 질문을 묻고 궁극적으로 답하도록 고안되었습니다. Center for Astrophysics는 매사추세츠주 케임브리지에 본사를 두고 있으며 미국 전역과 전 세계에 연구 시설을 갖추고 있습니다.

.First Complete Human Genome Improves DNA Sequencing and Strengthens Genetic Analysis
최초의 완전한 인간 게놈은 DNA 시퀀싱을 개선하고 유전자 분석을 강화합니다

주제:통풍유전학국립표준기술원 작성자 : NIST(NATIONAL INSTITUTE OF STANDARDS AND TECHNOLOGY)
2022년 4월 7일 유전자 분석 개념 우리의 유전자 코드를 구성하는 30억 개 이상의 문자를 완전히 철자하기 위해 오랜 공백을 채우는 새로 업데이트된 인간 게놈과 함께 별도의 동반 연구에서는 이것이 DNA 시퀀싱 능력 을 향상시키는 정확한 템플릿 역할을 할 수 있음을 보여주었습니다.
-도약과 한계. NIST(National Institute of Standards and Technology), 존스 홉킨스 대학교, 캘리포니아 대학교 데이비스가 이끄는 Telomere-to-Telomere(T2T) 컨소시엄 내 그룹 - 게놈 완성 계획 수천 명의 DNA 시퀀싱을 지원합니다. 사이언스 저널에 발표된 새로운 논문에서 연구원들은 게놈의 이전 표현으로 인해 생성된 수만 개의 오류를 수정했으며 200개 이상의 의학적 관련 유전자 분석에 더 우수하다는 것을 발견했습니다. 이 발견은 T2T의 게놈이 유전적 장애에 대한 연구를 크게 추진할 수 있고, 더 나아가 환자가 보다 신뢰할 수 있는 진단의 이점을 얻을 수 있음을 시사합니다.
-임상의와 연구원이 유전적 장애를 연구하거나 진단하기 위해 DNA의 염기서열을 분석할 때, 그들은 각각 환자 또는 연구 대상의 게놈의 한 부분을 미러링하는 일련의 DNA를 생성하는 기계를 사용합니다. 그런 다음 해당 문자열을 참조 게놈이라고 하는 템플릿과 비교하여 해당 문자열을 배치할 순서를 파악합니다. “DNA 시퀀싱이 퍼즐을 맞추는 것과 같다면 참조 게놈은 완성된 퍼즐 상자의 그림과 같습니다.
-연구의 공동 저자인 NIST 생물의학 엔지니어 Justin Zook은 이렇게 말했습니다. T2T 버전 이전의 가장 진보된 참조 게놈은 게놈의 8%가 부족하고, 과거 시퀀싱 기술로 해독하기 어려웠던 특정 섹션은 오류로 가득 차 있습니다. 이러한 불완전함으로 인해 빈칸이 있고 잘못된 위치에 조각이 표시되는 퍼즐 상자 그림과 유사합니다. 그러나 지난 20년 동안 유전체학에서 이루어진 기술적, 과학적 발전 덕분에 T2T 컨소시엄은 인간 참조 게놈을 채우고 정리할 수 있었습니다. Zook과 다른 연구 저자들은 완성된 참조가 DNA 시퀀싱에서 얼마나 많은 차이를 만들 수 있는지 보여주는 것을 목표로 했습니다.
팀 은 4개 대륙에서 온 수천 명의 사람들로부터 유전적으로 다양한 게놈 서열을 축적한 국제적 노력인 1000 Genomes Project(1KGP) 에서 참조에 대한 증명 기반을 찾았습니다 . 처음부터 시작하여 새로운 피험자로부터 DNA를 얻는 대신 연구원들은 1KGP에서 이미 배치된 DNA 세그먼트를 함께 조각할 수 있었습니다.
저자는 컴퓨터 프로그램을 사용하여 T2T 참조로 3,202개의 게놈을 분석하고 결과를 이전 참조로 수행된 이러한 게놈에 대한 출판된 작업과 비교했습니다. 두 참조 중 하나를 사용하여 결합된 게놈은 중요한 영역에서 크게 다르다는 것이 분명해졌습니다. T2T 참조 게놈은 수백만 개의 유전적 변이(사람마다 다른 DNA 스트레치)를 다른 참조가 하지 못한 것을 밝혀냈습니다. 또한 잘못 배치된 변형과 같은 수만 개의 흠집을 연속적으로 씻어냈습니다. 즉, 퍼즐 상자 그림의 빈칸을 채우는 새로운 변형과 수정을 통해 이전에는 수천 개가 어울리지 않았던 올바른 퍼즐 조각을 보여주었다.
“우리가 발견한 것은 이 새로운 레퍼런스가 전반적으로 정확도 를 개선했다는 것입니다. 따라서 개인의 혈통이 아프리카, 백인 또는 아시아인이든 상관없이 새로운 참조는 결과를 개선했습니다.”라고 Zook이 말했습니다. 새로운 참조의 능력을 더 철저하게 이해하기 위해 연구자들은 질병과의 연관성이 알려지거나 의심되는 269개 유전자의 변이를 식별하기 위해 이를 사용하려고 시도했습니다. 이 유전자는 이전에 정확하게 해독하기 어려웠던 게놈 영역에 숨겨져 있습니다.
저자들은 이 테스트를 수행하기 위해 수천 명이 아니라 NIST 주도 Genome in the Bottle Consortium으로 광범위하게 특징지어지는 한 사람에게 초점을 좁혔습니다 . 그들은 새로운 참조에 의해 뒷받침되는 일련의 강력한 시퀀싱 기술을 사용하여 유전자 코드 공개에 동의한 이 개인의 게놈에 대한 엄격한 분석을 수행했다고 Zook은 말했습니다.
그들의 노력으로 그들은 시퀀싱 방법을 평가할 때 답의 열쇠로 작용할 수 있는 게놈 벤치마크(관심 있는 유전자의 DNA에 대한 매우 정확한 디지털 판독)를 얻었습니다. 팀은 참조를 각각 세 가지 다른 시퀀싱 기술과 짝지었습니다. 그러나 접근 방식에 관계없이 T2T의 게놈은 항상 이전보다 성능이 뛰어났으며 하나의 기술로 오류를 최대 12배까지 줄였습니다.
T2T 참조 게놈은 우리의 유전 청사진 매핑을 완성하여 게놈 분야에서 중추적인 이정표를 표시합니다. 이 분야의 연구원들은 이제 과거에는 불가능했던 게놈 영역을 탐색하고 수많은 유전자가 다양한 질병과 어떻게 관련되는지 이해하기 시작할 수 있습니다. 그러나 Zook에 따르면 클리닉에서 이를 실행하기 전에 해야 할 일이 더 있습니다.
지금까지의 모든 표시에 따르면 T2T 기준이 현재 기준보다 더 정확합니다. 그러나 연구자들은 수백만 개의 게놈을 분석하기 위해 현재 참조를 사용하여 이를 사용할 때 결과를 적절하게 해석하는 데 필수적인 깊은 지식을 얻었습니다. 전문가들은 앞으로 나아가기 위해 같은 방식으로 새로운 참조의 내용을 파악해야 합니다. Zook은 "이 참조가 현재 액세스할 수 있는 게놈 영역에 있는 많은 개인의 DNA 서열의 정확성을 이해하기 위해서는 확실히 더 많은 작업이 있을 것이라고 생각합니다."라고 말했습니다.
관련 연구: 인간 게놈의 첫 번째 완전한 시퀀스에서 밝혀진 숨겨진 영역 과학자들은 마침내 완전한 인간 게놈을 시퀀싱하고 새로운 유전 비밀을 밝혀냈습니다 참조: Sergey Aganezov, Stephanie M. Yan, Daniela C. Soto, Melanie Kirsche, Samantha Zarate, Pavel Avdeev, Dylan J. Taylor, Kishwar Shafin, Alaina Shumate, Chunlin Xiao의 "완전한 참조 게놈은 인간 유전 변이 분석을 향상시킵니다." , Justin Wagner, Jennifer McDaniel, Nathan D. Olson, Michael EG Sauria, Mitchell R. Vollger, Arang Rhie, Melissa Meredith, Skylar Martin, Joyce Lee, Sergey Koren, Jeffrey A. Rosenfeld, Benedict Paten, Ryan Layer, Chen-Shan Chin, Fritz J. Sedlazeck, Nancy F. Hansen, Danny E. Miller, Adam M. Phillippy, Karen H. Miga, Rajiv C. McCoy, Megan Y. Dennis, Justin M. Zook 및 Michael C. Schatz, 2022년 4월 1일 , 과학 . DOI: 10.1126/science.abl3533
========================
메모 2204090631 나의 사고실험 oms스토리텔링
나는 오래전 부터 DNA의 염기서열이 샘플a.oms의 베이스 마방진을 닮았다고 비유하곤 했다. 인간의 염기 서열이 30억개이라 하던가? 그중에서도 유용한 유전자는 3만여개? 왜 이런 비율이 나타날까? 그 이유는 의미없어 보이는 배열 때문이라는데 그것은 정크 DNA가 아니다. 빈공간이 있는 배열조차도 의미가 있다는 뜻은 마방진이나 oms을 연구해보면 금새 이해가 된다. 허허.
그러한 빈 공간이나 의미없이 반복되는 배열이 있어야만 전체적으로 균형과 조화와 그리고 질서가 있는 마방진이 성립되고 oms,oss가 논리적으로 단순명확하게 조건적으로 성립된다. 우주에 빈공간이 왜 그리도 많겠나? 암흑 물질이나 암흑에너지가 있다고 하지만 아직도 그 물질의 정체를 아는가?
그 이유는 잘 모르지만 전체적으로 magicsum 아닌가? 전체적으로 시공간이 존재하면 그것이 존재의 그자체이고 질서이며 균형이고 조화이다. 하나 보태기 하나가 둘이 잖아? 하나 보태기 없음은 하나이고..그처럼 자연스런 빈공간은 존재하는 것이고 자연스런 존재1에 존재1은 두개의 시공간의 존재일 뿐이다. 철학이네! 허허.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
0000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):

- Leaps and limits. A group within the Telomere-to-Telomere (T2T) consortium led by the National Institute of Standards and Technology (NIST), Johns Hopkins University, and University of California Davis - The Genome Completion Plan supports the DNA sequencing of thousands. In a new paper published in the journal Science, the researchers corrected tens of thousands of errors generated by previous representations of the genome and found it superior to the analysis of more than 200 medically relevant genes. This finding suggests that the genome of T2T could greatly drive research into genetic disorders, and furthermore, patients could benefit from a more reliable diagnosis.
-When clinicians and researchers sequence DNA to study or diagnose a genetic disorder, they each use a machine that generates a series of DNA that mirrors a portion of the patient's or study subject's genome. It then compares that string to a template called a reference genome to determine the order in which to place the string. “If DNA sequencing is like putting a puzzle together, then the reference genome is like a picture of a finished puzzle box.
-NIST biomedical engineer Justin Zook, co-author of the study, said: The most advanced reference genomes before the T2T version lack 8% of the genome, and certain sections that have been difficult to decipher with past sequencing techniques are full of errors. These imperfections make it similar to a puzzle box picture with blanks and misplaced pieces. However, thanks to technological and scientific advances in genomics over the past two decades, the T2T consortium has been able to populate and clean up the human reference genome. Zook and other study authors aimed to show how much difference a finished reference can make in DNA sequencing.
===========================
memo 2204090631 my thought experiment oms storytelling
For a long time, I used to liken the base sequence of DNA to resemble the base magic square of sample a.oms. Did you say that humans have 3 billion nucleotide sequences? Among them, about 30,000 useful genes? Why do these ratios appear? The reason is because of the seemingly meaningless arrangement, which is not junk DNA. If you study magic square or oms, you will quickly understand that even an arrangement with empty space has meaning. haha.
Only when there is such an empty space or meaningless and repeated arrangement, a magic square with balance, harmony, and order is established as a whole, and oms, oss are logically and simply and conditionally established. Why is there so much empty space in the universe? There is dark matter or dark energy, but do you still know the identity of the matter?
I don't know why, but isn't it magicsum overall? If space-time exists as a whole, it is existence itself, order, balance, and harmony. One plus one equals two, right? One and no addition is one.. Such a natural empty space exists, and existence 1 in natural existence 1 is only the existence of two space-times. Philosophy! haha.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):
.Peering Into the Inner Workings of Evolution With Jellyfish Eyes
해파리의 눈으로 진화의 내부 작동 들여다보기
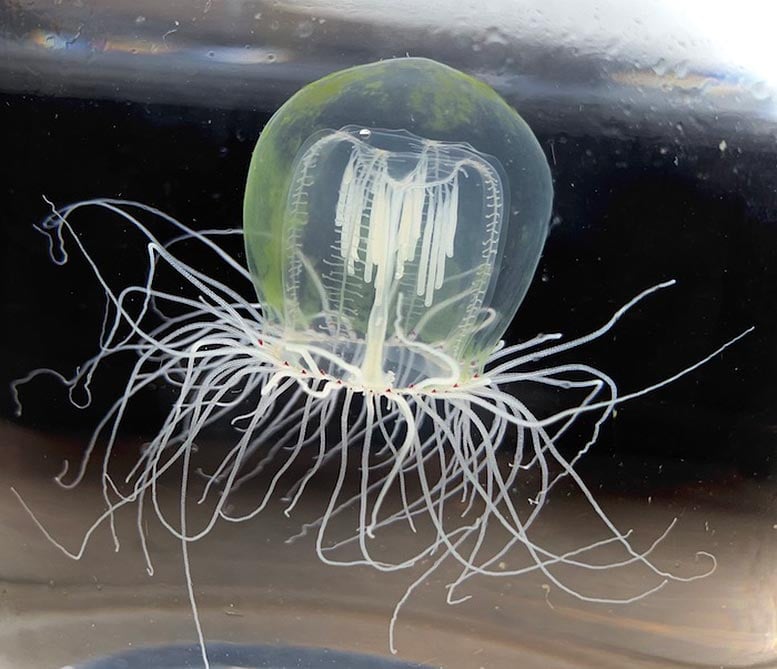
주제:진화해파리캔자스 대학교 캔자스 대학교 2022년 4월 8 일 레드 아이드 해파리 Polyorchis pencillatus라고 불리는 이 작은 해파리는 red-eyed jellyfish라고 합니다. 오첼리라고 불리는 여러 개의 눈은 촉수의 바닥에서 볼 수 있으며 빛을 감지하는 세포와 붉은 색소를 포함합니다. 크레딧: Anna Klompen
-일부 해파리는 눈이 단순합니다. 일부는 복잡한 것을 가지고 있습니다. 다른 해파리는 눈이 전혀 없습니다. 실제로, 최근 연구에 따르면 다른 종의 해파리 눈은 수천 년에 걸쳐 여러 번 다른 방식으로 개별적으로 독립적으로 진화하여 형질이 유전적으로 어떻게 표현되는지 더 잘 이해할 수 있는 이상적인 모델이 되었습니다. 이제 캔자스 대학의 생태 및 진화 생물학 교수인 Paulyn Cartwright가 포함된 연구원 팀; 텍사스 A&M 대학의 Maria Pia Miglietta, Galveston; UC 산타바바라의 수석 연구원인 Todd Oakley는 생명의 역사에서 중복된 사건인 해파리의 눈 "수렴"이 어떻게 진화가 작동하는지에 대한 창을 제공하는 방법을 연구하기 위해 국립 과학 재단으로부터 보조금을 받았습니다. 유전적, 세포적, 형태학적 수준. NSF 보조금의 KU 부분은 총 $494,890입니다.
-"눈은 해파리 내에서 독립적으로 여러 번 진화했습니다."라고 Cartwright는 말했습니다. "우리는 모든 동물의 눈이 하나의 기원이 아니라는 것을 한동안 알고 있었지만 해파리에서 눈이 독립적으로 진화했다는 사실에 놀랐습니다." 이제 Cartwright와 그녀의 협력자들은 해파리가 진화할 때마다 눈을 만들기 위해 유전 툴킷의 동일한 측면 또는 다른 측면을 사용하는지 여부를 발견하기 위해 진화 패턴에 대한 과학적 "심층 분석"을 계획합니다. 연구자들은 해파리 눈의 진화 역사를 추적하기 위해 Medusozoa(Cnidaria phylum의 분기군)의 상세한 계통수를 재구성하고 비교 전사체 및 단일 세포 RNA 시퀀싱을 사용하여 수렴 눈의 유전 경로의 유사점과 차이점을 결정할 계획입니다.
-"해파리는 빛을 감지하는 세포의 단순한 클러스터에서 매우 복잡한 눈에 이르기까지 다양한 눈을 가지고 있기 때문에 이에 대해 정말 좋은 시스템입니다. 이미지를 형성하고 렌즈, 각막 및 망막을 가질 수 있는 카메라 유형의 눈 — 표면적으로는 척추동물과 동일하게 보일 수 있습니다.”라고 카트라이트는 말했습니다. 그녀의 KU 연구실에서 그녀는 여러 종의 해파리를 분석하여 서로 다른 해파리 눈의 단일 세포에서 발현되는 모든 유전자를 결정하고 다양한 해파리 개체 간에 공유되는 것과 기본 유전자 구성 요소에서 변경된 사항을 찾을 계획입니다.
고려대 연구원은 “세포 자체에 고유한 특성이 있고 실제로는 다양한 유전자가 발현된 결과이기 때문에 개별 유전자를 보면 전체 패턴을 놓치는 경우가 있다”고 말했다. “그러나 우리가 세포에서 발현되는 모든 유전자와 그 특정 결과가 무엇인지 살펴본다면, 그것은 우리에게 다른 수준의 정보를 줄 수 있습니다. 그렇기 때문에 서로 다른 모든 수준을 살펴보고 이 매우 복잡한 질문을 이해하는 데 실제로 도움이 되도록 유사점과 변경된 사항을 확인하는 것이 좋습니다.
해파리는 이러한 유형의 실험에 매우 순응하기 때문에 이를 수행할 수 있는 훌륭한 시스템입니다. 개별 유전자와 발현 방식을 볼 수 있습니다. 우리는 그 세포에서 발현되는 모든 유전자를 전체적으로 볼 수 있습니다. 새로운 NSF 보조금에 따른 작업의 일부에는 표본을 수집하기 위해 해파리 생물다양성의 핫스팟인 파나마로의 여행이 포함될 것입니다. Cartwright와 그녀의 동료들은 연구 결과를 사용하여 해파리의 보다 상세한 계통 발생 수 또는 진화 역사를 구성할 것입니다. "해파리는 매우 다양합니다. 수천 종이 있습니다."라고 Cartwright는 말했습니다.
“이러한 다양성의 많은 부분이 5억 년 전에 일어났기 때문에 그들의 정확한 진화 역사를 밝히는 것은 부분적으로 정말 어려운 일이었습니다. 다른 과제는 이러한 유기체를 샘플링하는 것입니다. 그들 중 많은 수가 심해의 탁 트인 바다 환경에 살고 있으며 일부는 믿을 수 없을 정도로 작고 찾기가 어렵습니다. 그래서 우리는 다양하고 샘플링하기 어려운 그룹 내에서 고대 발산 시간을 보고 있습니다. 이것은 제 경력의 최소 지난 20년 동안 작업해 온 도전이었습니다. 그래서 우리는 더 많은 데이터를 얻고 더 많은 유전자를 시퀀싱할 수 있고 더 많은 DNA 를 던질 수 있기 때문에 유전체학 시대에 정말 흥분됩니다.이 문제의 시퀀스는 Cnidarians 간의 이러한 관계 중 일부를 해결하는 데 매우 유망할 것으로 예상됩니다."
https://scitechdaily.com/peering-into-the-inner-workings-of-evolution-with-jellyfish-eyes/
========================
메모 2204090603 나의 사고실험 oms스토리텔링
해파리의 눈은 단순하기도하고 아예 눈이 없다고도 한다. 그런데 어떤 연구는 상자해파리들이 위쪽 맹그로브(mangrove) 나무에 초점을 유지하기 위해 그들의 눈을 사용하고 있음을 발견했다. 원시 신경계를 가진 한 동물에서 이러한 발달된 시각 능력의 발견은 놀라운 것이다. 단순한 생물의 감각계가 매우 복잡한 적응을 어떻게 사용하게 되었는지는 이해되지 않고 있다. 8개의 눈은 척추동물(vertebrates)의 눈을 닮았고 4가지의 유형의 24개의 눈을 가졌다는 연구도 있다.
원시동물이 어떻게 눈을 가질 수 있을까? 이는 샘플a.oms는 해파리이다. '어떻게 촉수=1의 값을 가지는가? '하는 문제처럼 들린다. 본시 자연에는 'oms.eye'가 있다. 수천억개을 가진 원시 눈이다. 생명체이든 아니든 그것은 oms.eye이다. 허허. 빛이 눈이다. 무한한 빛살은 oms.eye를 가졌다. 시공간을 순간적으로 바라본다. 소설쓰네! 허허.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
0000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):

-"The eye has evolved many times independently within the jellyfish," Cartwright said. "We've known for a while that the eyes of all animals aren't of one origin, but we're surprised that eyes evolved independently in jellyfish." Now, Cartwright and her collaborators plan a scientific "deep analysis" of evolutionary patterns to discover whether each time a jellyfish evolves, it uses the same or different aspects of its genetic toolkit to make its eyes. To trace the evolutionary history of the jellyfish eye, the researchers plan to reconstruct the detailed phylogenetic tree of Medusozoa (a clade of the Cnidaria phylum) and use comparative transcriptome and single-cell RNA sequencing to determine similarities and differences in the genetic pathways of the converging eye.
-"This is a really good system for this because jellyfish have eyes that range from simple clusters of light-sensing cells to very complex eyes. Camera-type eyes that can shape images and have lenses, corneas and retinas — Superficially, they can look identical to vertebrates,” Cartwright said. In her KU lab, she analyzed several species of jellyfish to determine all the genes expressed in a single cell in different jellyfish eyes, and to determine the number of genes expressed in different jellyfish populations. We plan to look for what is shared between the livers and what has changed in the underlying genetic components.
- Scientists have known for more than a century that box jellyfish (also known as cubozoans) have a unique array of eyes. Box jellyfish have 4 different types of eyes, a total of 24 eyes.
The eight eyes resemble those of vertebrates. The eyes “have a sophisticated lens, retina, iris, cornea, etc., all contained within one eye only 0.1 mm in diameter.” [2] However, the characteristics of their function and performance have not been explored. It was only known that the eyes provided the box jellyfish with sufficient vision to respond to light and shadow and to avoid obstacles. (The box jellyfish doesn't just float in the water and eat anything it catches on its tentacles, but rather swim towards objects of interest and turn out to be active predators that can dodge others).
https://creation.kr/animals/?idx=1291162&bmode=view
===========================
Memo 2204090603 My Thought Experiment oms Storytelling
Jellyfish eyes are often simple, and it is said that they do not have eyes at all. However, some studies have found that box jellyfish are using their eyes to maintain focus on the upper mangrove trees. The discovery of such a developed visual ability in an animal with a primitive nervous system is surprising. It is not understood how the sensory systems of simple creatures came to use very complex adaptations. Eight eyes resemble those of vertebrates, and some studies have shown that they have 24 eyes of four types.
How could primitive animals have eyes? This sample a.oms is a jellyfish. 'How do you get a tentacle=1 value? ' sounds like a problem. In nature, there is 'oms.eye'. It is a primitive eye with hundreds of billions. Living or not, it is oms.eye. haha. light is the eye Infinite beams have oms.eye. Looking at space-time in an instant. Write a novel! haha.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.quasi oms(standard)
0100000010=0,2
0010000100
0001000001
0010001000
0001010000
0000100100
0000100010
2000000000
0000001001
sample b.prime oms(standard)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
=bigrip/zerosum, npir+c(dark energy)
sample c.oss
domain(2203080543):
댓글