.Our DNA is becoming the world's tiniest hard drive
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
http://jk0620.tripod.com
https://www.facebook.com/junggoo.lee.9

.Supercomputers reveal how X chromosomes fold, deactivate
슈퍼 컴퓨터는 X 염색체가 어떻게 접히고 비활성화되는지 보여줍니다
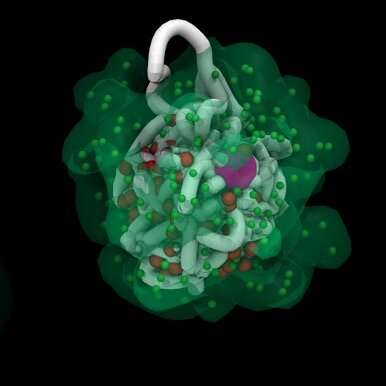
로 로스 알 라모스 국립 연구소 RNA 입자는 X 염색체 비활성화의 새로운 시각화에서 마우스의 X 염색체를 떼지었습니다. 크레딧: 로스 알라모스 국립 연구소 OCTOBER 4, 2021
실험 데이터를 기반으로 한 슈퍼컴퓨터 기반 동적 모델링을 사용하여 연구자들은 이제 암컷 포유류 배아에서 하나의 X 염색체를 끄는 과정을 조사할 수 있습니다. 이 새로운 기능은 생물학자들이 X 비활성화 과정에서 RNA의 역할과 염색체의 구조를 이해하는 데 도움이 되며, 이는 유전자 발현에 대한 더 깊은 이해로 이어지며 유전자 기반 장애 및 질병에 대한 약물 치료에 대한 새로운 경로를 열어줍니다.
로스 알라모스 국립 연구소의 방문 과학자이자 매사추세츠 종합 병원 및 하버드 부서의 고분자 물리학자인 Anna Lappala는 "염색체 주위에 퍼져 있는 모든 RNA를 모델링하고 이를 차단할 수 있었던 것은 이번이 처음"이라고 말했습니다. 분자 생물학. Lappala는 Proceedings of the National Academy of Sciences 10월 4일자에 발표된 논문의 첫 번째 저자입니다 . "2D이고 정적인 실험 데이터만으로는 이 정도의 세부 사항에서 전체 염색체를 볼 수 있는 해상도가 없습니다. 이 모델링을 통해 유전자 발현을 조절하는 프로세스를 볼 수 있으며 모델링은 2D에 기반을 두고 있습니다.
Massachusetts General Hospital과 Harvard의 공동 작업자가 제공한 실험 데이터입니다." 4차원으로 시간을 포함하여 움직임을 보여주기 때문에 4D로 간주되는 이 모델은 Los Alamos 슈퍼컴퓨터에서 실행됩니다. 이 모델은 또한 4DHiC라는 분자 방법을 통해 얻은 마우스 게놈의 실험 데이터를 통합합니다. 결합된 분자 및 계산 방법론은 최초입니다. 시각화에서 RNA 입자는 X 염색체 위에 떼를 지어 있습니다. 얽힌 스파게티 모양의 가닥이 몸을 비틀면서 모양이 바뀌면 입자가 염색체의 깊숙한 곳을 집어삼키고 침투하여 꺼집니다.
https://youtu.be/qM5jOaU-55U
시각화 보기: "이 방법은 우리가이 후생 유전 학적 과정의 상호 작용 모델을 개발할 수 있습니다,"누구의 실험실 출자 지니 T. 리, 매사추세츠 종합 병원의 분자 생물학에서 하버드 의과 대학에서 유전학의 교수 및 부의장 말했다 실험 데이터 모델을 뒷받침는. 후성유전학은 게놈의 돌연변이를 포함하지 않는 유전자 발현 및 유전 형질의 변화에 대한 연구입니다.
Lee는 "현장에서 누락된 것은 컴퓨터에 능숙하지 않은 사용자가 양방향으로 염색체에 들어갈 수 있는 방법입니다."라고 말했습니다. 그녀는 Los Alamos 모델을 사용하는 것과 "X 염색체의 모든 위치를 확대하고 좋아하는 유전자를 선택 하고 주변 의 다른 유전자 를 보고 상호 작용하는 방식을 볼 수 있는" Google Earth를 사용하는 것과 비교 했습니다.
그 능력은 예를 들어 질병이 어떻게 퍼지는지에 대한 통찰력을 제공할 수 있다고 그녀는 말했습니다. 로스 알라모스 국립 연구소의 구조 생물학자인 카리사 산본마츠(Karissa Sanbonmatsu)는 이 논문의 작업을 기반으로 로스 알라모스는 현재 모든 과학자가 게놈 데이터를 업로드하고 이를 3D로 다양한 배율로 동적으로 볼 수 있는 Google 어스 스타일 브라우저를 개발하고 있다고 말했습니다.
해당 논문의 교신저자이자 계산 방법을 개발하는 프로젝트 리더입니다. 포유류에서 여성 배아는 각 부모로부터 하나씩 유전되는 2개의 X 염색체로 잉태됩니다 . X 비활성화는 배아가 생존하는 데 중요한 단계인 염색체를 차단하고 X 비활성화의 변화는 다양한 발달 장애를 유발할 수 있습니다. 새로운 Los Alamos 모델은 다양한 유전자 기반 질병 및 장애에 대한 약리학적 치료로 이어질 수 있는 유전자 발현 및 관련 문제에 대한 더 깊은 이해를 촉진할 것이라고 Lee는 말했습니다.
-Sanbonmatsu는 "우리의 주요 목표는 염색체의 모양이 변하는 것을 확인하고 시간이 지남에 따라 유전자 발현 수준을 확인하는 것이었습니다."라고 말했습니다. Sanbonmatsu는 유전자가 어떻게 켜지고 꺼지는지 이해하기 위해 "염색체의 구조를 아는 것이 정말 도움이 됩니다.
가설은 압축되고 단단히 구조화된 염색체가 유전자를 끄는 경향이 있지만 흡연 총은 많지 않습니다. 움직이는 3D 구조를 모델링함으로써 우리는 구조적 압축과 유전자 끄기 사이의 관계에 더 가까이 다가갈 수 있습니다." Lee는 염색체의 구조를 종이접기에 비유했습니다.
-종이접기 학과 유사한 복잡한 모양은 유전자 발현을 위한 많은 표면을 제공하며 활성 상태를 유지하는 것이 생물학적으로 선호될 수 있습니다. 이 모델은 염색체의 다양한 하위 구조를 보여줍니다. 폐쇄되면 "일부 하부 구조는 유지되지만 일부는 해체되는 단편적인 프로세스"라고 Sanbonmatsu는 말했습니다. "우리는 점진적인 전환을 통해 시작, 중간 및 끝 단계를 봅니다. 우리가 후성 유전적 변화에서 상세한 구조적 전환을 분석할 수 있었던 것은 이번이 처음이기 때문에 후성 유전학에 중요합니다." 모델링은 또한 X 염색체 비활성화를 피하는 염색체 표면의 유전자를 보여 초기 실험 작업을 확인합니다.
모델에서 그들은 클러스터링되고 염색체 표면에서 분명히 상호 작용하거나 함께 작동합니다. 모델링의 또 다른 통찰력에서 "염색체가 여전히 상당히 클 때 활성 X에서 작은 비활성 X로 이동함에 따라, 우리는 극도로 조밀한 염색체 코어가 있음을 알 수 있지만 표면은 훨씬 적습니다. 우리는 표면에서도 훨씬 더 많은 움직임을 볼 수 있습니다."라고 Lappala가 말했습니다. "그렇다면 염색체가 재배열될 수 있는 너무 빠르거나 느리지 않은 중간 영역이 있습니다." 비활성 X는 나중에 비활성 X의 연령 관련 활성화라고 하는 과정에서 활성화될 수 있습니다. "이는 특히 자가면역을 유발하는 것으로 알려진 혈액 세포의 문제와 관련이 있습니다."라고 Lee는 말했습니다. "일부 연구는 활성 X 염색체에 없는 것을 되돌려줌으로써 어린이의 신경 장애를 치료하기 위해 비활성 X를 활성화하기 위해 약리학적으로 시도하고 있습니다. 예를 들어, 어린이는 질병을 일으킬 수 있는 돌연변이를 가질 수 있습니다. 우리는 우리가 다시 활성화할 수 있다면 비활성 X에 정상적인 사본이 있다면 우리는 그 돌연변이에 대한 후성적 치료를 받게 될 것입니다."
추가 탐색 연구는 X 염색체의 구조에 대한 새로운 단서를 보여줍니다 추가 정보: 4D 염색체 재구성은 포유류 X 염색체 PNAS (2021) 의 시공간 재구성을 설명합니다 . doi.org/10.1073/pnas.2107092118 저널 정보: 국립과학원 회보 에 의해 제공 로스 알 라모스 국립 연구소
https://phys.org/news/2021-10-supercomputers-reveal-chromosomes-deactivate.html
====================
메모 211005824 나의 사고실험 oms 스토리텔링
슈퍼 컴퓨터는 X 염색체가 어떻게 접히고 비활성화되는지 보여주었다. 종이접기 염색체는 샘플2. oss의 베이스(magic square)의 모습이다. 압축되고 구조화되어 oss을 만나 제2의 베이스(2배}를 2^43개로 증식 시켰다.
그리고, 제2의 베이스를 다시 매개로한 제100만의 베이스에서는 그 종이접기는 블랙홀 중력을 1그램안에 접혀놓을 수 있음이여. 허허.
Sample 1. 12th oms
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample 2/oss
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

-Sanbonmatsu said, "Our main goal was to see changes in the shape of chromosomes and to determine the level of gene expression over time." "Knowing the structure of chromosomes is really helpful," Sanbonmatsu said to understand how genes are turned on and off.
The hypothesis is that compressed, tightly structured chromosomes tend to turn off genes, but not many smoking guns. By modeling the moving 3D structure, we can come closer to the relationship between structural compression and gene turn-off." Lee compared the structure of a chromosome to an origami.
======================
memo 211005824 my thought experiment oms storytelling
The supercomputer showed how the X chromosome folds and becomes inactive. Origami chromosomes are sample 2. A view of the base (magic square) of oss. Compressed and structured, it met oss and multiplied the second base (doubled) to 2^43.
And, in the 1 millionth base with the second base again as a medium, the origami can fold the black hole's gravity within 1 gram. haha.
Sample 1. 12th oms
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample 2/oss
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

.Our DNA is becoming the world's tiniest hard drive
우리의 DNA는 세계에서 가장 작은 하드 드라이브가 되고 있습니다
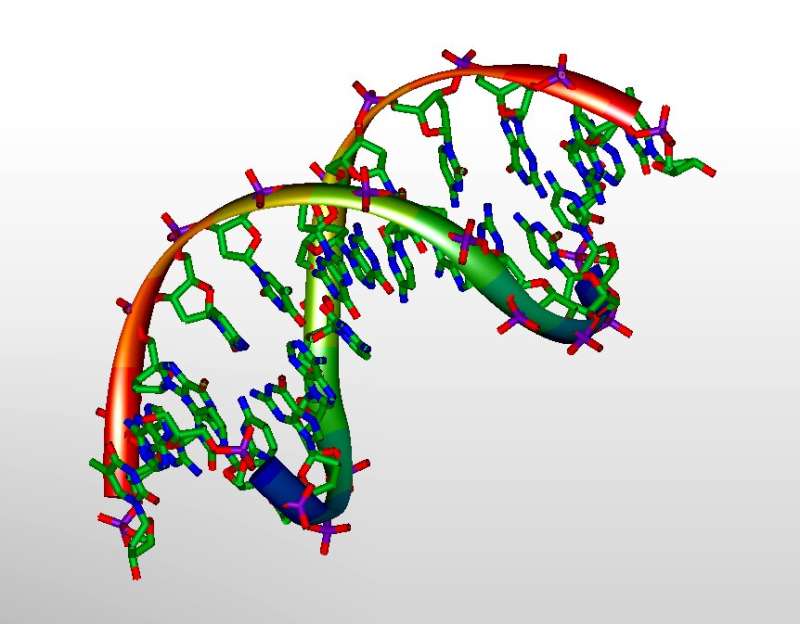
Lila Reynolds, Northwestern University DNA의 3D 모델. 출처: Michael Ströck/Wikimedia/ GNU 무료 문서 라이선스 OCTOBER 4, 2021
-우리의 유전자 코드는 비용이 많이 들고 엄청난 양의 에너지와 공간을 사용하는 기존 솔루션보다 데이터를 저장하는 데 수백만 배 더 효율적입니다. 사실, 우리는 하드 드라이브를 없애고 지구상의 모든 디지털 데이터를 수백 파운드의 DNA 안에 저장할 수 있습니다.
-DNA를 고밀도 데이터 저장 매체로 사용하는 것은 바이오센싱 및 바이오레코딩 기술과 차세대 디지털 저장 분야에서 획기적인 발전을 이룰 수 있는 잠재력을 가지고 있지만, 연구자들은 이 기술을 확장할 수 있는 비효율성을 극복하지 못했습니다. 이제 Northwestern University의 연구원들은 완료하는 데 몇 시간 또는 며칠이 아닌 몇 분이 걸리는 DNA에 정보를 기록하는 새로운 방법을 제안합니다.
-연구팀은 새로운 효소 시스템을 사용하여 빠르게 변화하는 환경 신호를 직접 DNA 서열로 기록하는 DNA를 합성했는데, 이 방법은 과학자들이 뇌 내부의 뉴런을 연구하고 기록하는 방식을 바꿀 수 있다고 이 논문의 수석 저자는 말했습니다.
"효소적 DNA 합성을 사용하여 분 단위로 시간 신호를 기록하는" 연구는 목요일(9월 30일) Journal of the American Chemical Society에 게재되었습니다 . 이 논문의 수석 저자인 노스웨스턴 엔지니어링 교수인 Keith EJ Tyo는 그의 연구실이 데이터 저장을 위한 새로운 솔루션을 만들기 위해 DNA의 자연적 능력을 활용하는 데 관심이 있다고 말했습니다.
-Tyo는 "자연은 DNA를 복사하는 데 능숙하지만 우리는 DNA를 처음부터 쓸 수 있기를 정말로 원했습니다."라고 말했습니다. "이를 수행하는 생체 외(신체 외부) 방법은 느린 화학적 합성을 포함합니다. 우리의 방법은 DNA를 합성하는 효소가 직접 조작될 수 있기 때문에 정보를 작성하는 데 훨씬 저렴합니다. 최첨단 세포 내 기록은 모두 미리 발현되고 지속적으로 정보를 저장할 수 있는 우리의 효소와 달리 신호에 대한 반응으로 단백질 발현의 기계적 단계가 필요하기 때문에 훨씬 느립니다."
McCormick 공과대학의 화학 및 생물 공학 교수인 Tyo는 Center for Synthetic Biology의 회원으로 미생물과 환경 변화를 감지하고 신속하게 대응하는 메커니즘을 연구합니다. 단백질 발현 우회 세포 내 분자 및 디지털 데이터를 DNA에 기록하는 기존 방법은 기존 DNA 서열에 새로운 데이터를 추가하는 다중 부분 프로세스에 의존합니다. 정확한 기록을 생성하기 위해 연구자는 특정 단백질의 발현을 자극하고 억제해야 하며, 완료하는 데 10시간 이상이 소요될 수 있습니다.
-Tyo 연구실은 Tdt for Local Environmental Signals 또는 TURTLES를 사용한 Time-Sensitive Untemplated Recording이라는 새로운 방법을 사용하여 템플릿을 복사하는 대신 완전히 새로운 DNA를 합성하여 더 빠르고 더 높은 해상도로 기록할 수 있다고 가정했습니다. DNA 중합효소가 염기를 계속 추가 함에 따라 환경 변화가 합성하는 DNA의 구성에 영향을 미치기 때문에 데이터가 몇 분 단위로 유전 코드 에 기록됩니다 .
금속 농도의 변화와 같은 환경 변화는 중합효소에 의해 기록되어 "분자 티커 테이프"로 작용하여 과학자들에게 환경 변화의 시기를 알려줍니다. DNA에 변화를 기록하기 위해 바이오센서를 사용하는 것은 세포 내부에서 거북이의 생존 가능성을 증명하는 주요 단계를 나타내며, 연구자들이 기록된 DNA를 사용하여 뉴런이 서로 통신하는 방법을 배울 수 있는 능력을 제공할 수 있습니다.
-공동 제1저자이자 Tyo 연구소의 박사후 연구원인 Namita Bhan은 "이것은 언젠가 수백만 개의 세포 사이의 상호작용을 동시에 연구할 수 있는 방법에 대한 정말 흥미로운 개념 증명입니다."라고 말했습니다. "이전에 보고된 직접 효소 변조 기록 시스템은 없다고 생각합니다." 뇌세포에서 오염된 물까지 확장성과 정확성에 대한 더 많은 잠재력을 갖춘 TURTLES는 뇌 연구를 발전시키는 도구의 기반을 제공할 수 있습니다.
Tyo 연구실의 공동 제1저자이자 대학원생이기도 한 Alec Callisto에 따르면, 연구자들은 오늘날의 기술로 뇌 뉴런의 아주 작은 부분만을 연구할 수 있으며, 그렇다고 해도 그들이 알고 있는 것에는 한계가 있습니다. 과학자들은 뇌의 모든 세포 내부에 레코더를 배치함으로써 많은 (백만) 뉴런에 걸쳐 단일 세포 해상도로 자극에 대한 반응을 매핑할 수 있었습니다. 칼리스토는 "현재 기술이 시간이 지남에 따라 확장되는 방식을 보면 기존 기술과 동시에 바퀴벌레의 뇌 전체를 기록할 수 있기까지 수십 년이 걸릴 수 있습니다. 인간 뇌에 있는 수백억 개의 뉴런은 고사하고요."라고 말했습니다.
"그래서 그것은 우리가 정말로 가속화하고 싶은 것입니다." 외부에서 TURTLES 시스템은 데이터 저장 요구의 폭발적인 증가(2025년까지 최대 175제타바이트)를 해결하기 위해 다양한 솔루션에 사용될 수도 있습니다. 이는 팀에서 "한 번 쓰고 절대 읽지 않는" 데이터라고 하지만 사고가 발생할 경우 액세스할 수 있어야 하는 데이터라고 부르는 폐쇄 회로 보안 영상을 저장하는 것과 같은 장기 보관 데이터 애플리케이션에 특히 좋습니다.
엔지니어가 개발한 기술을 통해 수년간 사랑받는 카메라 메모리를 저장하는 하드 드라이브 및 디스크 드라이브도 DNA 조각으로 대체될 수 있습니다. 저장 외에도 "티커 테이프" 기능을 바이오센서로 사용하여 음용수의 중금속 농도와 같은 환경 오염 물질을 모니터링할 수 있습니다. 실험실이 디지털 및 셀룰러 녹음 모두에서 개념 증명을 넘어서는 데 초점을 맞추는 동안 팀은 더 많은 엔지니어가 개념에 관심을 갖고 연구에 중요한 신호를 기록하는 데 사용할 수 있기를 희망한다고 밝혔습니다. Tyo는 "우리는 여전히 강력한 세포 내 기록에 필요한 게놈 인프라와 세포 기술을 구축하고 있습니다"라고 말했습니다. "이것은 우리의 장기 목표에 도달하기 위한 한 단계입니다."
추가 탐색 나노전극은 수천 개의 연결된 포유동물 뉴런을 내부에서 기록합니다. 추가 정보: Namita Bhan et al, Enzymatic DNA Synthesis를 사용하여 분 분해능으로 시간 신호 기록 , 미국 화학 학회 저널 (2021). DOI: 10.1021/jacs.1c07331 저널 정보: Journal of the American Chemical Society 노스웨스턴대학교 제공
https://phys.org/news/2021-10-dna-world-tiniest-hard.html
====================
메모 2110050620 나의 사고실험 oms 스토리텔링
DNA가 정보에 훌륭한 저장매체이라고 한다. 인간은 자신의 지적 정보를 자신의 DNA에 쉽게 저장하는 방법을 개발할 것이다. 그리고 그 DNA를 유전 시키면 아기들도 그 정보로 이미 천재성을 태어나자 마자 발휘하게 된다. 허허.
우주 정보의 무한대 저장매체 개발이 필요한가? 샘플2. oss가 그 기회를 제공할 것이다. 그리고 샘플1. oms의 smola_d 구조의 얽힘이동이 격자 다층복잡계 네트워크를 형성하여 우주의 안팎의 그 모든 정보를 1초이내 처리할거여.
Sample 1. 12th oms
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample 2/oss
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

-Our genetic code is millions of times more efficient at storing data than conventional solutions that are expensive and use huge amounts of energy and space. In fact, we could get rid of hard drives and store all the digital data on the planet within hundreds of pounds of DNA.
-Using DNA as a high-density data storage medium has the potential to make breakthroughs in biosensing and biorecording technologies and next-generation digital storage, but researchers have not overcome the inefficiencies that make this technology scalable. Now, researchers at Northwestern University are proposing a new way to write information into DNA that takes minutes rather than hours or days to complete.
The team used a novel enzyme system to synthesize DNA that records rapidly changing environmental signals directly as DNA sequences, a method that could change the way scientists study and record neurons inside the brain, the paper's lead author said.
The study "Using Enzymatic DNA Synthesis to Record Time Signals in Minutes" was published Thursday (September 30) in the Journal of the American Chemical Society. Northwestern engineering professor Keith EJ Tyo, lead author of the paper, says his lab is interested in harnessing the natural power of DNA to create novel solutions for data storage.
-Co-first author and postdoctoral fellow at the Tyo Institute, Namita Bhan, said, "This is a really exciting proof of concept how one day we can study the interactions between millions of cells simultaneously." "I don't think there are any direct enzyme-modulated recording systems reported before," she said. With more potential for scalability and accuracy, from brain cells to contaminated water, TURTLES could provide the foundation for tools that advance brain research.
======================
memo 2110050620 my thought experiment oms storytelling
DNA is said to be an excellent storage medium for information. Humans will develop an easy way to store their intellectual information in their DNA. And if you inherit that DNA, babies will already show their genius as soon as they are born with that information. haha.
Is it necessary to develop an infinite storage medium for space information? sample 2. oss will provide that opportunity. and sample 1. The entangled movement of the smola_d structure of oms forms a lattice multi-layered complex system, which will process all the information inside and outside the universe within 1 second.
Sample 1. 12th oms
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample 2/oss
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
댓글