.Astronomers Discover Binary System Ejecting Common Envelope at 450,000 MPH
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
http://jk0620.tripod.com
https://www.facebook.com/junggoo.lee.9
.Astronomers Discover Binary System Ejecting Common Envelope at 450,000 MPH
천문학자, 450,000 MPH에서 공통 봉투를 방출하는 이진 시스템 발견

주제:천문학천체물리학중국과학원 중국과학원 2022년 7월 14 일 이진 시스템의 공통 엔벨로프 진화 이진법 J 1920에서 공통 봉투 진화에 대한 예술가의 인상. 제공: Jingchuan Yu
중국과 호주 천문학자들은 이진 공통 엔벨로프 진화에 대한 직접적인 증거를 찾습니다 천문학자 팀은 초당 약 200km(450,000마일)의 속도로 공통 봉투를 방출하는 쌍성계를 공동으로 발견했습니다. 연구진은 중국과학원의 윈난천문대(YAO)의 Zhanwen Han 교수와 호주국립대학교 의 Chris Wolf 교수가 주도했습니다 . 천문학자들이 쌍성 진화의 핵심 과정인 공통 봉투 진화의 직접적인 증거를 관찰한 것은 이번이 처음입니다.
이 중요한 발견은 관측을 통해 쌍성계의 일반적인 봉투 진화를 정확하게 특성화하는 방법을 제공합니다. 이 연구는 2022년 7월 7일 Royal Astronomical Society의 Monthly Notices에 게재되었습니다 . 별은 우주에서 빛나는 물체의 대부분을 구성합니다. 모든 별의 절반 이상이 쌍성계의 일부라고 믿어집니다. 쌍성계의 두 별은 중력에 의해 서로의 주위를 공전합니다. 항성 물체의 운명은 쌍성계의 진화에 크게 영향을 받습니다. 이것은 Ia형 초신성, 이중 블랙홀 , 이중 중성자별 등 을 포함한 특이한 항성 물체의 형성과 같은 천문학 및 천체 물리학의 대부분의 신비에 대한 설명으로 자주 인용되었습니다 .

Binary Star의 일반적인 봉투 진화 쌍성 진화에서 일반적인 봉투 진화의 그림. 크레딧: YAO 공통 봉투
-진화는 이진 진화의 핵심 프로세스 중 하나입니다. 이 과정에서 쌍성계의 도너 별은 질량 손실로 인해 극적으로 팽창하여 두 별이 서로를 향해 나선형으로 회전하고 공통 봉투가 형성됩니다. 이 프로세스는 이진 시스템의 후속 진화를 결정합니다. 공통 봉투가 성공적으로 방출되면 더 짧은 궤도 주기를 가진 쌍성계가 형성될 것으로 예상됩니다. 그렇지 않으면 공통 봉투에 있는 두 개의 별이 단일 개체로 병합됩니다. 일반 봉투는 1976년 B. Paczynski에 의해 처음 가정되었습니다. 그러나 일반 봉투는 지금까지 본 적이 없습니다. 결과적으로 과학자들은 쌍성 진화의 공통 엔벨로프 단계에서 정확히 무슨 일이 일어나는지에 대해 상당히 불분명했습니다.
호주 국립 대학교 의 2.3미터 광시야 망원경 과 케플러 우주 망원경 의 관측을 바탕으로 중국과 호주 과학자들은 J 1920이라는 이름 의 뜨거운 하위 왜성과 백색 왜성 으로 구성된 쌍성계를 공동으로 발견했습니다. 이 쌍성에서 시스템에서 두 별은 약 3.5시간의 공전 주기로 서로를 공전하며 점점 더 가까워지고 있습니다. 또한 과학자들은 이 쌍성계가 초당 약 200km(450,000마일)의 속도로 움직이는 팽창하는 껍질로 둘러싸여 있음을 확인했습니다.
이 팽창하는 껍질은 약 10,000년 전에 쌍성계에서 배출된 일반적인 봉투로 더 확인되었습니다. J 1920 쌍성계에서 관측된 지속적인 궤도 수축은 외피에 있는 두 별의 궤도 운동으로 인한 마찰이 궤도 각운동량을 심각하게 분산시킬 수 있음을 나타냅니다. 이것은 자기 제동 및 중력 복사의 메커니즘 외에도 각운동량 손실에 대한 새로운 메커니즘입니다. 이 중요한 발견의 의의는 이론적 아이디어를 현실로 바꾼다는 것입니다. 과학자들은 공통 외피 진화의 첫 번째 증거를 보았을 뿐만 아니라 관찰을 통해 쌍성별의 공통 외피 진화를 정확하게 특성화할 수 있었습니다.
참조: Jiangdan Li, Christopher A Onken, Christian Wolf, Péter Németh, Mike Bessell, Zhenwei Li, Xiaobin Zhang, Jiao Li의 "로슈 로브를 채우는 뜨거운 하위 왜성 및 백색 왜성 바이너리: 배출된 공통 봉투의 가능한 감지?", Luqian Wang, Lifang Li, Yangping Luo, Hailiang Chen, Kaifan Ji, Xuefei Chen 및 Zhanwen Han, 2022년 7월 7일, Royal Astronomical Society의 월간 공지사항 DOI : 10.1093/mnras/stac1768
https://scitechdaily.com/astronomers-discover-binary-system-ejecting-common-envelope-at-450000-mph/
===================
메모 2207150534 나의 사고실험 oms 스토리텔링
우주의 쌍성계에는 봉투모델이 있다. 이과정을 샘플c.oss처럼 피드백화 시키면 별들이 무척 복잡하게 배치된 은하계를 설명할 수 있다. 수많은 별들이 결국 샘플c.oss.공유봉투 원리의 급진적 진화처럼 보인다.
진화는 이진 진화의 핵심 프로세스 중 하나이다. 이 과정에서 쌍성계의 도너 별은 질량 손실로 인해 극적으로 팽창하여 두 별이 서로를 향해 나선형으로 회전하고 공통 봉투가 형성된다. 이 프로세스는 이진 시스템의 후속 진화를 결정한다. 공통 봉투가 성공적으로 방출되면 더 짧은 궤도 주기를 가진 쌍성계가 형성될 것으로 예상된다. 그렇지 않으면 공통 봉투에 있는 두 개의 별이 단일 개체로 병합된다. 일반 봉투는 1976년 B. Paczynski에 의해 처음 가정되었다. 그러나 일반 봉투는 지금까지 본 적이 없다. 결과적으로 과학자들은 쌍성 진화의 공통 엔벨로프 단계에서 정확히 무슨 일이 일어나는지에 대해 상당히 불분명했다.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.qoms(standard)
0000000011=2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000
0010000001
sample b.poms(standard)
p&pp=6n-1(+1)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

-Evolution is one of the key processes of binary evolution. During this process, the donor star of the binary system expands dramatically due to the loss of mass, causing the two stars to spiral towards each other and a common envelope is formed. This process determines the subsequent evolution of binary systems. If the common envelope is successfully released, a binary system with shorter orbital periods is expected to form. Otherwise, two stars in a common envelope are merged into a single object. The ordinary envelope was first hypothesized by B. Paczynski in 1976. But I've never seen an ordinary envelope before. As a result, scientists have been fairly obscure about what exactly happens during the common envelope phase of binary evolution.
=====================
memo 2207150534 my thought experiment oms storytelling
There is an envelope model in the binary system of the universe. If this process is fed back like sample c.oss, it can explain the galaxy in which the stars are very complex. Numerous stars eventually appear to be a radical evolution of the sample c.oss. common envelope principle.
Evolution is one of the key processes of binary evolution. During this process, the donor star of the binary system expands dramatically due to the loss of mass, causing the two stars to spiral towards each other and a common envelope is formed. This process determines the subsequent evolution of the binary system. A successful release of the common envelope is expected to form a binary system with a shorter orbital period. Otherwise, two stars in a common envelope are merged into a single entity. The ordinary envelope was first hypothesized by B. Paczynski in 1976. But I've never seen an ordinary envelope before. As a result, scientists have been fairly obscure about what exactly happens in the common envelope stage of binary evolution.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.qoms(standard)
0000000011=2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000
0010000001
sample b.poms(standard)
p&pp=6n-1(+1)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

.Physicists use AI to find the most complex protein knots so far
물리학자들은 AI를 사용하여 지금까지 가장 복잡한 단백질 매듭을 찾습니다
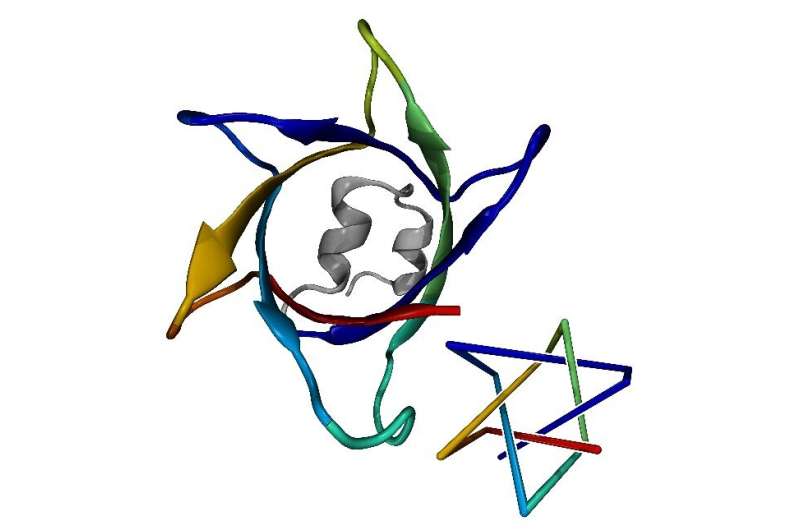
요하네스 구텐베르크 대학 마인츠 AlphaFold가 예측한 7개의 교차점(왼쪽)과 단순화된 표현(오른쪽)이 있는 가장 복잡한 단백질 매듭. 크레딧: Maarten Brems JULY 14, 2022
-단백질의 화학적 구성(아미노산 서열)이 어떻게 3차원 구조를 결정하는지에 대한 질문은 반세기 이상 동안 생물 물리학에서 가장 큰 도전 과제 중 하나였습니다. 소위 단백질의 "접힘"에 대한 이러한 지식은 무엇보다도 다양한 질병 및 치료에 대한 이해에 크게 기여하기 때문에 수요가 많습니다. 이러한 이유로 Google DeepMind 연구팀은 3D 구조를 예측하는 인공 지능 AlphaFold를 개발했습니다. Johannes Gutenberg University Mainz(JGU)와 University of California, Los Angeles의 연구원으로 구성된 팀은 이제 이러한 구조를 자세히 살펴보고 매듭과 관련하여 조사했습니다.
우리는 주로 신발 끈과 케이블에서 매듭을 알고 있지만 세포의 나노 규모에서도 발생합니다. 매듭이 있는 단백질은 구조 예측 의 품질을 평가하는 데 사용할 수 있을 뿐만 아니라 접는 메커니즘과 단백질의 진화에 대한 중요한 질문을 제기합니다.
-AlphaFold에 대한 테스트로 가장 복잡한 매듭 "우리는 새로운 단백질 매듭 에 대한 AlphaFold의 모든 예측(약 100,000개)을 수치적으로 조사했습니다 ."라고 Ph.D.인 Maarten A. Brems가 말했습니다. 마인츠 대학 Peter Virnau 박사 그룹의 학생. 목표는 AlphaFold의 예측에 대한 실험적 검증을 위한 기초를 제공하기 위해 이전에 알려지지 않은 복잡하고 단백질 매듭을 포함하는 희귀한 고품질 구조를 식별하는 것이었습니다.
-이 연구는 지금까지 가장 복잡한 매듭이 있는 단백질을 발견했을 뿐만 아니라 단백질에서 처음으로 복합적인 매듭을 발견했습니다. 후자는 동일한 끈에 있는 두 개의 개별 매듭 으로 생각할 수 있습니다. "이러한 새로운 발견은 또한 그러한 희귀한 단백질 뒤에 있는 진화적 메커니즘 에 대한 통찰력을 제공합니다 ."라고 이 프로젝트에 참여한 이론 물리학자인 로버트 런켈(Robert Runkel)은 덧붙였습니다.
이 연구 결과는 최근 Protein Science 에 게재되었습니다 . Peter Virnau 박사는 결과에 만족합니다. "우리는 이미 UCLA의 Todd Yeates 박사와 협력하여 이러한 구조를 실험적으로 확인했습니다. 이 연구 라인은 인공 지능에 대한 생물 물리학 커뮤니티의 관점을 형성할 것이며 우리는 운이 좋습니다. Todd Yeates와 같은 전문가를 참여시키는 것입니다."
추가 탐색 물리학자들은 실험적인 3차원 염색체 구조를 조사하기 위해 수학적 알고리즘을 사용합니다.
추가 정보: Maarten A. Brems et al, AlphaFold는 가장 복잡한 단백질 매듭 및 복합 단백질 매듭 예측, Protein Science (2022). DOI: 10.1002/pro.4380 John Jumper et al, AlphaFold를 사용한 매우 정확한 단백질 구조 예측, Nature (2021). DOI: 10.1038/s41586-021-03819-2 저널 정보: 네이처 Johannes Gutenberg University Mainz 제공
https://phys.org/news/2022-07-physicists-ai-complex-protein.html
===================
메모 2207150500 나의 사고실험 oms 스토리텔링
샘플c.oss의 베이스는 마방진이다. 이는 단백질의 접힘을 함의한다. 고차원 구조로 표현되는 접힘은 2차원에 있을 뿐이다. 생명의 진화에 단백질의 접힘의 구조적 해석이 필요한 실정이다. 이를 인공지능이 답을 주고 있는 모양이다. 허허.
인공지능 AlphaFold에 대한 테스트로 가장 복잡한 매듭 혹은 새로운 단백질 매듭 에 대한 AlphaFold의 모든 예측(약 100,000개)을 수치적으로 조사했다. 이 연구는 지금까지 가장 복잡한 매듭이 있는 단백질을 발견했을 뿐만 아니라 단백질에서 처음으로 복합적인 매듭을 발견했다. 후자는 동일한 끈에 있는 두 개의 개별 매듭 으로 생각할 수 있다. 이러한 새로운 발견은 또한 그러한 희귀한 단백질 뒤에 있는 진화적 메커니즘 에 대한 통찰력을 제공한다 .
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.qoms(standard)
0000000011=2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000
0010000001
sample b.poms(standard)
p&pp=6n-1(+1)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca

-The question of how a protein's chemical makeup (amino acid sequence) determines its three-dimensional structure has been one of the greatest challenges in biophysics for more than half a century. This knowledge of the so-called "folding" of proteins is in demand, among other things, because it greatly contributes to the understanding of various diseases and treatments. For this reason, the Google DeepMind research team developed AlphaFold, an artificial intelligence that predicts 3D structures. A team of researchers from Johannes Gutenberg University Mainz (JGU) and the University of California, Los Angeles has now taken a closer look at these structures and investigated them in relation to knots.
- Most complex knot as a test for AlphaFold "We numerically examined all of AlphaFold's predictions (about 100,000) for novel protein knots," said Ph.D. Maarten A. Brems. Student of the Dr. Peter Virnau group at the University of Mainz. The goal was to identify rare, high-quality structures containing previously unknown complex and protein knots to provide a basis for experimental validation of AlphaFold's predictions.
-This study not only found the most complex knotted protein to date, but also found the first complex knotted protein in a protein. The latter can be thought of as two separate knots on the same string. "These new discoveries also provide insight into the evolutionary mechanisms behind such rare proteins," adds Robert Runkel, a theoretical physicist who worked on the project.
=====================
memo 2207150500 my thought experiment oms storytelling
The base of sample c.oss is magic square. This implies folding of the protein. Folds represented by high-dimensional structures are only in two dimensions. Structural interpretation of protein folding is necessary for the evolution of life. It seems that artificial intelligence is giving the answer. haha.
As a test for the AI AlphaFold, we numerically examine all of AlphaFold's predictions (about 100,000) for the most complex knots or new protein knots. This study not only found the most complex knotted protein to date, but also found the first complex knot in a protein. The latter can be thought of as two separate knots on the same string. These new discoveries also provide insight into the evolutionary mechanisms behind such rare proteins.
Sample a.oms (standard)
b0acfd 0000e0
000ac0 f00bde
0c0fab 000e0d
e00d0c 0b0fa0
f000e0 b0dac0
d0f000 cae0b0
0b000f 0ead0c
0deb00 ac000f
ced0ba 00f000
a0b00e 0dc0f0
0ace00 df000b
0f00d0 e0bc0a
sample b.qoms(standard)
0000000011=2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000
0010000001
sample b.poms(standard)
p&pp=6n-1(+1)
q0000000000
00q00000000
0000q000000
000000q0000
00000000q00
000000000q
0q000000000
000q0000000
00000q00000
0000000q000
000000000q0
sample c.oss(standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
zybzzfxzy
cadccbcdc
cdbdcbdbb
xzezxdyyx
zxezybzyy
bddbcbdca
댓글