.Molecular atlas of spider silk production could help bring unparalleled material to market
http://blog.naver.com/mssoms
http://jl0620.blogspot.com
http://jk0620.tripod.com
https://www.facebook.com/junggoo.lee.9
.Molecular atlas of spider silk production could help bring unparalleled material to market
거미줄 생산의 분자 아틀라스는 비교할 수 없는 재료를 시장에 출시하는 데 도움이 될 수 있습니다

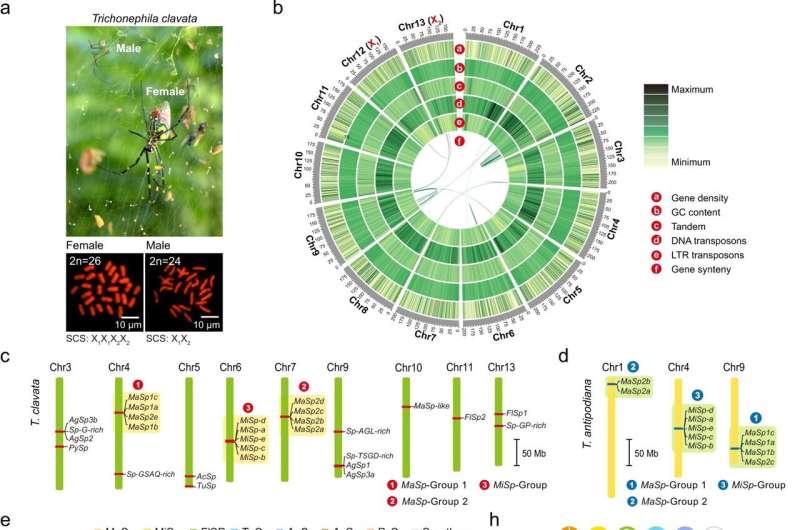
저스틴 잭슨, Phys.org T. clavata의 염색체 규모 게놈 어셈블리 및 전체 spidroin 유전자 세트. 성체 암컷과 성체 수컷을 황금색 구줄(위)과 암컷 및 수컷 핵형(아래)에서 보여주는 T. clavata 의 사진 . SCS, 성 염색체 시스템. b 13개의 슈도염색체( Mb 척도에서 Chr1 – 13) 의 게놈 환경을 나타내는 원형 다이어그램 . c 염색체에 고정된 28개의 T. clavata spidroin 유전자. d 다른 orb-web 거미, T. antipodiana 의 Spidroin 유전자 그룹 . 공개된 T. antipodiana 의 게놈 데이터spidroin 유전자의 위치 정보를 확인하기 위해 분석되었습니다. e 6개의 orb-web 거미 종의 Spidroin 유전자 카탈로그. f 견사선(주요 및 소부폭(Ma 및 Mi), 편모형-(Fl), tubuliform-(Tu), 집합체-(Ag) 및 aciniform & pyriform(Ac & Py) 땀샘) 및 독샘의 발현 클러스터링. 분홍색 선은 Ma와 Mi 땀샘 사이의 가장 가까운 관계를 보여줍니다. g T. clavata 견사선 의 형태 . 3개의 독립적인 실험에서 유사한 결과가 얻어졌으며 소스 데이터에 요약되었습니다. h 서로 다른 유형의 견사선에서 28가지 spidroin 유전자의 발현 패턴. 소스 데이터는 소스 데이터 파일로 제공됩니다. 신용: 자연 커뮤니케이션(2023). DOI: 10.1038/s41467-023-36545-6 MARCH 3, 2023
중국 사우스웨스트 대학교(Southwest University)의 연구원들은 특히 강한 황금색 거미줄로 알려진 황금 구체를 짜는 거미인 트리코네필라 클라바타(Trichonephila clavata)의 전체 염색체 규모 게놈 어셈블리와 완전한 스피드로인 유전자 세트를 구성했습니다. 그들은 그들의 작업이 "거미 드래그라인 실크 생성에 대한 지식을 크게 확장하는 다차원 데이터를 제공합니다..."라고 증명하고 연구원들은 거미가 거미줄을 제조하는 방법을 더 잘 이해하기 위해 이 새로운 "분자 지도"를 사용할 계획입니다.
Nature Communications 저널에 게재된 이 논문은 야생 거미 포획에서 다중 분석에 이르기까지 연구자들이 취한 단계를 자세히 설명하여 드래그라인 실크 생산을 담당하는 거미의 주요 팽대선 내 유전자의 상호작용을 밝혀냈습니다. 스파이더 드래그라인 실크는 잠재적인 의료 및 산업 응용 분야가 많은 진정한 경이로운 소재입니다. 강철보다 가볍고 강하면서도 고무에 필적하는 탄성 신축성을 유지합니다. 많은 합성 물질 과 달리 거미줄은 독성이 없고 생분해되며 생체 적합성이 있어 외과용 임플란트, 바이오센서 및 조직 재건에 이상적인 물질입니다.
현재 우리가 사용하는 많은 재료 목록을 대체하기 위해 거미줄을 채택하는 데 있어 유일한 제한은 제조가 얼마나 어려운가입니다. 예전에 Nexia라는 회사에서 산양유에 들어 있는 단백질을 생산하려는 노력이 있었고, 효과가 있었지만 대량 생산에 필요한 규모는 아니었습니다. 그리고 거미줄의 명백한 이점에도 불구하고 필요한 규모로 거미 양식을 시작하려는 사람은 아무도 없습니다.
-연구원들은 거미의 분자 수준에서 거미줄 생산을 더 잘 이해함으로써 이 비할 데 없는 물질을 시장에 출시하는 데 도움이 되는 실용적인 통찰력을 얻을 것으로 기대합니다. 멀티오믹스로 분자 지도 만들기 게놈을 얻기 위해 연구팀은 모든 유전자 시퀀서 중 가장 확장된 연속 읽기를 생성할 수 있는 Oxford Nanopore 플랫폼과 더 정확하지만 더 짧은 읽기 캡처 길이를 위한 Illumina 시퀀싱 기계 및 염색체 매핑을 위한 Hi-C를 사용했습니다. 이 세 가지 다른 게놈 데이터 세트를 결합함으로써 연구원들은 거미의 염색체 규모 게놈 조립 및 완전한 spidroin 유전자 세트의 상세한 모델을 생물정보학적으로 재구성할 수 있었습니다.
-이 게놈 데이터를 가지고 있으면 유전자 발현 과 궁극적으로 거미줄에서 발견되는 단백질 사이의 연결이 가능해집니다 . 이것이 바로 연구원들이 다음에 한 일입니다. 연구팀은 주요 팽창샘의 세 부분에 대한 전사체(메신저 RNA), 단백질 및 대사체(신호 분자) 분석을 수행했습니다. 꼬리, 주머니 및 덕트. 액체 크로마토그래피-질량 분석 결과 28개의 단백질이 확인되었습니다. 10개는 거미줄을 구성하는 단백질인 스피드로인, 15개는 거미줄 구성 요소, 1개는 독과 관련이 있습니다.
핵심 구성 요소가 확인되면 연구자들은 강도 기반 절대 정량화 순서로 순위를 매길 수 있습니다. 추가 분석을 통해 유전자 및 유전자 산물의 기능을 기반으로 실크 생산 과 관련된 Tail, Sac 및 Duct의 특정 생물학적 기능을 특성화할 수 있었습니다 . Tail omics는 주로 유기산 합성을 중심으로 진행되었으며 Sac의 오믹스는 주로 지질 생산에 중점을 두었고 Duct omics는 이온 교환 및 키틴 합성과 관련되었습니다. 이전 연구는 현재 연구에서 발견된 몇 가지 요소를 발견했지만 그 어떤 것도 전체 그림을 그렇게 완전하고 포괄적인 방식으로 통합하지 못했습니다.
추가 정보: Wenbo Hu 외, 분자 아틀라스는 거미 드래그라인 실크의 3단 회전 메커니즘을 밝힙니다, Nature Communications (2023). DOI: 10.1038/s41467-023-36545-6 저널 정보: Nature Communications © 2023 사이언스엑스네트워크
https://phys.org/news/2023-03-molecular-atlas-spider-silk-production.html
==============================
메모 2303040637 나의 사고실험 oms 스토리텔링
생물학적인 단백질의 능력들이 생물마다 다양한 능력을 가지고 있다. 연구원들은 특히 강한 황금색 거미줄로 알려진 황금 구체를 짜는 거미인 Trichonephila clavata의 전체 염색체 규모 게놈 어셈블리와 완전한 스피드로인 유전자 세트를 구성했다. 거미가 거미줄을 제조하는 방법을 더 잘 이해하기 위해 이 새로운 "분자 지도"를 사용할 계획이란다.
놀라운 능력을 가진 단백질들이 생물들의 생존방식으로 진화되어 갔다. 이곳에는 늘 샘플링 oss.proteins.base가 존재한다. 치밀하고 빈틈이 없는 분자지도가 단순히 분자배열일 뿐인데 자연적인 능력을 나타낸다. 어떻게 그런 일이 가능할까? 자연은 일종에 magissum으로 연결된 분자지도를 통해 생명의 구조체를 만들어 놓은 것이다.
어떻게? 예를들어 분자의 지도가 샘플링 oss.base에서 마방진이 나타난다고 가정해보자. 그러면 그어떤 원소들도 빈틈없이 그 위치에 역할을 수행해야 한다. 가로의 몇째줄에 분자배열이 틀려서 거미줄의 능력이 oms.전방위적.자유자재적 탁월한 거미의 능력이 상실될 수 있는 문제이다. 허허.

Researchers at Southwest University, China, have organized a set of all -chromosomes of Triconephila Clavata, a golden spider known as a strong golden spider. They prove that their work provides a multidimensional data that greatly expands the knowledge of the creation of spider dragline silk ... "and researchers have this new" molecular map "to better understand how spiders make spider webs. I plan to use it.
Researchers expect to better understand the production of spider web at the molecular level of spiders, which will help to help to launch this comparable substance to the market. To obtain a molecular map creation genome with multi -omics, the team has an Oxford Nanopore platform that can generate the most extended continuous reads among all gene sequencers, and Hi for Illumina sequencing machine and chromapping for more accurate but shorter reading capture length I used -c. By combining these three different genomic data sets, researchers were able to reorganize the detailed models of the spider's chromosome -scale genomic and complete SPIDROIN gene sets.
-The genomic data can be connected between gene expression and ultimate protein found in spider webs. This is what researchers do next. The team analyzed the transcript (messenger RNA), protein and metabolites (signal molecules) for three portions of the main expansion glands. Tails, pockets and ducts. Liquid chromatography-As a result of mass analysis, 28 proteins have been identified. Ten pieces are the proteins that make up the spider web, 15 are spider web components, and 1 is related to poison.
============================
Memo 2303040637 My Accident Experiment OMS Story Telling
Biological proteins have a variety of abilities for each creature. The researchers formed a set of entire chromosomes of Trichonephila Clavata, a golden spider known as a strong golden spider web, and a genetic set with a complete speed. We plan to use this new "molecular map" to better understand how spiders make a spider web.
Proteins with amazing abilities have evolved into the way of survival. There is always a sampling OSS.Proteins.base here. The molecular maps are only molecular arrangements, which show natural abilities. How can that happen? Nature is a kind of molecular map connected to Magissum.
how? For example, suppose that the map of the molecule appears in the sampling OSS.base. Then any element must play a role in that position. The molecular arrangement is wrong in the several lines of the horizontal lines, which is a problem that the ability of the spider web can be lost. haha.
Samplea.oms (Standard)
B0ACFD 0000E0
000AC0 F00BDE
0C0FAB 000e0d
E00D0C 0B0FA0
F000E0 B0DAC0
D0F000 CAE0B0
0b000f 0EAD0C
0DEB00 AC000F
CED0BA 00F000
A0b00e 0dC0F0
0ACE00 DF000B
0F00D0 e0bc0a
Sampleb. Qoms (Standard)
0000000011 = 2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000-mser.2
0000000011
Sample B. Poms (Standard)
Q0000000000
00Q00000000
0000Q000000
000000Q0000
00000000q00
0000000000q
0Q000000000
000Q0000000
00000q00000
0000000q000
000000000q0
Sample C.OSS (Standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
Zybzfxzy
CADCCBCDC
CDBDCBDBB
XZEZXDYYX
zxezybzyy
bddbcbdca

.New Technology Can Detect Cancer Cells Without Invasive and Expensive Surgery
침습적이고 값비싼 수술 없이 암세포를 탐지할 수 있는 신기술

주제:바이오센서생명 공학암기술 대학교 시드니 University OF TECHNOLOGY 시드니 2023년 3월 3일 정적 액적 미세유체 장치 Static Droplet Microfluidic 장치는 원발성 종양에서 떨어져 혈류로 들어간 순환하는 종양 세포를 신속하게 감지할 수 있습니다. 신용: Majid Warkiani 박사의 사진MARCH 3, 2023
암 발견 및 치료를 개선하기 위한 신기술 새로운 장치는 침습적이고 값비싼 수술 없이 암세포를 탐지할 수 있습니다. University of Technology Sydney(UTS)의 연구원들은 혈액 샘플에서 암세포를 감지하고 분석할 수 있는 새로운 장치를 개발하여 의사가 침습적 생검 수술을 피하고 치료 진행 상황을 모니터링할 수 있도록 했습니다. 암은 호주에서 질병과 사망의 주요 원인이며 매년 150,000명 이상의 호주인이 진단을 받고 있습니다. 특히 간, 결장 또는 신장과 같은 장기에 암이 의심되는 환자는 확정 진단을 위해 종종 수술이 필요합니다. UTS 의생명공학부 마지드 와르키아니 교수는 조직검사를 받는 것은 환자에게 불편함을 줄 수 있을 뿐만 아니라 수술과 높은 비용으로 인한 합병증 위험이 증가할 수 있지만 효과적인 치료를 위해서는 정확한 암 진단이 필수적이라고 말했다.
-“혈액 샘플의 종양 세포 평가를 통해 암을 관리하는 것은 조직 생검을 하는 것보다 훨씬 덜 침습적입니다. 이를 통해 의사는 반복 검사를 수행하고 치료에 대한 환자의 반응을 모니터링할 수 있습니다.”라고 그는 말했습니다. Static Droplet Microfluidic 장치는 원발성 종양에서 떨어져 혈류로 들어간 순환하는 종양 세포를 신속하게 감지할 수 있습니다. 이 장치는 암의 고유한 대사 신호를 사용하여 정상 혈액 세포와 종양 세포를 구별합니다. "Rapid metabolomic screening of cancer cells via high-throughput static droplet microfluidics"라는 제목의 이 연구는 상호 심사 과학 저널인 Biosensors and Bioelectronics 에 게재되었습니다 .
-“1920년대에 Otto Warburg는 암세포가 많은 양의 포도당을 소비하여 더 많은 젖산을 생성한다는 사실을 발견했습니다. 우리 장치는 세포 주변의 산성화를 감지하는 pH 민감성 형광 염료를 사용하여 증가된 젖산염에 대해 단일 세포를 모니터링합니다.”라고 Warkiani 교수는 말했습니다. “단지 1밀리리터의 혈액에 들어 있는 수십억 개의 혈구 중 하나의 종양 세포가 존재할 수 있어 찾기가 매우 어렵습니다.
-새로운 검출 기술에는 대사적으로 활성인 종양 세포의 수를 분리하고 분류할 수 있는 38,400개의 챔버가 있습니다.”라고 그는 말했습니다. 종양 세포가 장치로 식별되면 유전 및 분자 분석을 거쳐 암 진단 및 분류에 도움이 되고 개인화된 치료 계획을 알릴 수 있습니다. 순환하는 종양 세포는 또한 암이 먼 장기로 이동하는 전이의 전구체이며 암 관련 사망의 90%를 유발합니다.
-이러한 세포를 연구하면 암 전이의 생물학에 대한 통찰력을 얻을 수 있으며, 이를 통해 새로운 치료법을 개발할 수 있습니다. 기존의 액체 생검 기술은 시간이 많이 걸리고 비용이 많이 들며 숙련된 작업자에게 의존하기 때문에 임상 환경에서의 적용이 제한됩니다. 이 새로운 기술은 고급 장비와 숙련된 작업자에 의존하지 않고 연구 및 임상 실험실에 통합하도록 설계되었습니다. 이를 통해 의사는 실용적이고 비용 효율적인 방식으로 암 환자를 진단하고 모니터링할 수 있습니다.
UTS 연구팀은 Static Droplet Microfluidic 장치에 대한 잠정 특허를 출원했으며 제품을 상용화할 계획입니다. 참조: "Rapid metabolomic screening of cancer cells via high-throughput static droplet microfluidics" by Payar Radfar, Lin Ding, Laura Rodriguez de la Fuente, Hamidreza Aboulkheyr, David Gallego-Ortega 및 Majid Ebrahimi Warkiani, 2022년 12월 16일, Biosensors and Bioelectronics . DOI: 10.1016/j.bios.2022.114966
==============================
메모 2303040714 나의 사고실험 oms 스토리텔링
혈액검사를 통한 임상적으로 정상적인 세포와 암세포를 어떻게 구별할까? 정상적인 혈액의 움직임을 샘플링 oss.base을 통과하게 하여 magicsum의 값을 얻었다고 가정해보자. 의심이 들면 더 세밀히 암을 추적하는 단계적인 정상적 혈액여부의 과정을 무한히 진행할 수 있다.
아무튼 1차적으로 base에 입고된 검사용 혈액을 간단히 초기값 base로 비교할 수 있다. 1차 비교값에서 가장 인상적으로 암세포가 많은 양의 포도당을 소비하여 더 많은 젖산을 생성한다는 사실로 인하여, 세포 주변의 산성화를 감지하는 pH 민감성 형광 염료를 통해 증가된 젖산염에 대해 단일 세포를 모니터링하게 된다. 허허.

-“Management of cancer through tumor cell assessment of blood samples is much less invasive than tissue biopsy. This allows the doctor to perform a repeated test and monitor the patient's response to treatment, ”he said. The static droplet microfluidic device can quickly detect tumor cells that are circulating from the primary tumors to the bloodstream. The device uses a unique metabolic signal of cancer to distinguish between normal blood cells and tumor cells. This study, titled "Rapid Metabolomic Screening of Cancer Cells Via High-ThroughPut Static Droflet Microfluidics, is published in the Biosensors and Bioelectronics, a mutual screening journal.
In the 1920s, OTTO Warburg found that cancer cells produce more lactic acid by consuming large amounts of glucose. Our device uses a pH -sensitive fluorescent dyes that detect acidification around the cells to monitor single cells for increased lactic acid, ”said Warkiani. “It is very difficult to find because there are only one of the billions of blood cells in the blood of only one millilitator.
============================
Memo 2303040714 My Accident Experiment OMS Story Telling
How do you distinguish clinically normal and cancer cells through blood tests? Suppose you have gained the value of Magicsum by passing the normal blood movement through sampling OSS.base. If you are suspicious, you can endeavor the process of normal blood to track cancer more in detail.
In any case, the blood for the base in the base can be simply compared to the initial value base. The fact that cancer cells produce more lactic acid by consuming large amounts of glucose in the primary comparison value, and monitoring single cells for increased lactic acid through pH -sensitive fluorescent dye, which detects acidification around cells. do. haha.
Samplea.oms (Standard)
B0ACFD 0000E0
000AC0 F00BDE
0C0FAB 000e0d
E00D0C 0B0FA0
F000E0 B0DAC0
D0F000 CAE0B0
0b000f 0EAD0C
0DEB00 AC000F
CED0BA 00F000
A0b00e 0dC0F0
0ACE00 DF000B
0F00D0 e0bc0a
Sampleb. Qoms (Standard)
0000000011 = 2,0
0000001100
0000001100
0000010010
0001100000
0101000000
0010010000
0100100000
2000000000-mser.2
0000000011
Sample B. Poms (Standard)
Q0000000000
00Q00000000
0000Q000000
000000Q0000
00000000q00
0000000000q
0Q000000000
000Q0000000
00000q00000
0000000q000
000000000q0
Sample C.OSS (Standard)
zxdxybzyz
zxdzxezxz
xxbyyxzzx
Zybzfxzy
CADCCBCDC
CDBDCBDBB
XZEZXDYYX
zxezybzyy
bddbcbdca
댓글